垂直顯像SMI
垂直顯像SMI是1996年在SMI與SPDM的基礎上開發出來的,能在奈米等級上最快分析完整的3D細胞結構的光學顯微鏡,有效的奈米級光學解像度,在解析2D圖像能達到5 nm,而在解析3D圖像能達到40 nm,所以比起以Abbe定律所算出來的物理極限200 nm還要更佳。[1] 恩斯特·阿貝在1873年提出理論上光學顯微鏡的解像度限制假說。
垂直顯像SMI光學顯微鏡是由海德堡大學光學應用與資訊處理博士克里斯托夫克勒梅所開發出來,集結了定位光學顯微鏡(光學間距精密顯微鏡SPDM, Spectral Precision Distance Microscopy)結構照明設備(空間調整照明設備SMI, Spatially Modulated Illumination)的科技。
自從2008年3月起,許多標準的螢光染劑像是綠色熒光蛋白(GFP)與Alexa螢光染劑可以應用在SPDMphymod (可物理修飾螢光團physically modifiable fluorophores)定位光學顯微鏡上,這種顯微鏡只有單一雷射波長才有適合的光強度能用在奈米圖解上。

配置
[編輯]SMI是特別的雷射光學照明設備 (空間調整照明設備Spatially Modulated Illumination)與用Vertico反射垂直向的光,使之能夠分析固定住的標本細胞也能分析光學解像度在10奈米甚致更少的活細胞 (1奈米 = 1 nm = 1 × 10−9 m).
此項科技的特別之處與聚焦科技,例如4Pi顯微鏡相比,差在較寬視野能讓整個細胞能在奈米等級的解像度下完整的描繪出來。這種整個細胞的3D顯像技術在20 µm × 20 µm的範圍下,只需花2分鐘,寬視野的顯像能讓整個物體的照明與針測同時進行。
空間調整照明設備(SMI: Spatially Modulated Illumination)
[編輯]
SMI 光學顯微鏡是建立在點擴散函數工程的光學處理技術之上,用以修正顯微鏡的點分散函數(PSF) 來增加光學解像度,使之能以波長等級來測量螢光物質的距離,分析其他結構參數則能達到奈米等級。
SMI顯微鏡在海德堡大學已經達到下列成果: 每個物件照明的強度都不一樣,與傳統的寬視野螢光顯微鏡不同,而是以兩個相反方向的干涉雷射光來調整空間的精確性,這種空間上的調整波長原理是在1993 由Bailey et al發表。[2][3]
SMI可以與其他超解析科技結合,像是與3D LIMON或是LSI-TIRF側向顯像技術而成全內反射。SMI 科技能允許讀出自動螢光基團(autofluorophore)分佈在人類眼睛組織的光學圖像,這使用了3種不同激發波長(488、568 、647 nm)而能收集自動螢光發出的光譜訊號,這項技術已經應用在人類眼睛組織的黃斑部退化上。[4]
定位顯微鏡(SPDM: Localization Microscopy)
[編輯]
SPDM (光學間距精密顯微鏡Spectral Precision Distance Microscopy),定位光學顯微鏡的基礎是熒光顯微鏡加上光學處理使得能測量"光學上分離"粒子的位置、距離與角度(例如:分子)光學顯微鏡的解析限制之下。 "光學上分離"指的是在一定的時間之內的一點上, 只有單一粒子或是分子在特定的區域被傳統的光學解像度下(大概是直徑200-250 nm)偵測到。當所有的分子在某個區域都含有不同的光學標記就能實現(例如:不同的顏色或者不同粒子發射的不同激發光 )
使用SPDM能達到結構的解析且能偵測兩點間空間上的最短距離,位置則由粒子的不同光學特性定位(拓墣解析)。在合適的條件下,則是依定位的精確度, 粒子密度等等決定,"拓墣解析"則是對應"頻率偏移調變"。
SPDM 是一種定位顯微鏡能達到有效的光學解析較於傳統的光學解析(大概200-250 nm)更佳,能顯出有效點像函數的最大值的一半寬,藉由應用合適的雷射光學精密處理技術,位置與距離小於點分散函數(傳統上200-250 nm) 的一半寬,且能被偵測目標物與不同的光學訊號精確到奈米等級。[5] 其中一項重要的應用是在基因研究方面(研究基因組中有功能的部分),另一項則是研究膜的構造。
定位光學顯微鏡的其中一個里程碑是1996年第一次在3D奈米等級上定位螢光物質,[6]也因此從理論上與實驗上證明定位的精確度。[7][8]
SPDMphymod: GFP快門模式標準螢光染劑(Standard fluorescent dyes in the blinking mode like GFP)
[編輯]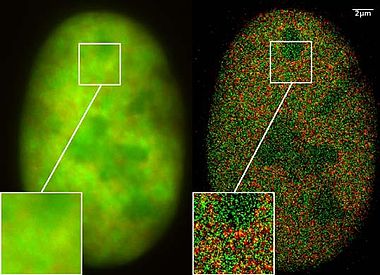
在過去兩年有分子應用在奈米顯微研究,此分子能射出一樣光譜頻率的光(但在光學的閃光特性上則不同)且能像光學間距精密顯微鏡一樣藉由光來調控能否射出光。能藉由結合上千張相同細胞的影像且使用雷射光譜精密測量來記錄定位的影像達到改善光學解像度。應用這種特別的奈米顯微處理技術在過去是非常困難的,因為過去以為只有特殊製造的分子才能使用光來調控能否射出光。
在2008年3月,克里斯托夫克勒梅的實驗室發現在某種光學-物理條件下也可以使用許多標準螢光染劑GFP、Alexa染劑與螢光素分子,使用SPDMphymod (可物理修飾螢光團physically modifiable fluorophores)技術,在適合的光強度下,單一雷射則可以顯出奈米級的影像。與其他需要兩條雷射的定位顯微鏡不同,使用特別的光-調控/光-活化螢光分子。[9]
GFP基因很早就被引進而且表現在許多原核與真核生物中,由於他們發現與開發GFP ,諾貝爾化學獎在2008年頒發給馬丁·查爾菲、下村脩與 錢永健。標準螢光分子除了能應用在SPMD上,還能應用在許多其他的領域上像是生物物理學, 細胞生物學與醫學等等。
SPDMphymod科技已經能使用標準螢光染劑:GFP, RFP, YFP, Alexa 488, Alexa 568, Alexa 647, Cy2, Cy3, Atto 488與螢光素。
奈米等級光學顯微鏡(LIMON): 3D超解析顯微鏡
[編輯]奈米等級光學顯微鏡(LIMON Light MicrOscopical nanosizing microscopy) 在2001年海德堡大學發明,同時結合定位顯微鏡與空間調整照明設備在3D超解析顯微鏡上。
結合SMI與SPDM使垂直顯像SMI能繪出3D圖像,因此第一次SMI計劃與SPDM計劃始動。SMI計劃決定粒子的中心與粒子的在顯微鏡軸的分散方向。然而粒子或分子的中心可以精確到1-2 nm,分散離中心的程度可以被由軸直徑精確到30-40 nm。
隨後,側向粒子或分子則採用SPDM來定位且能精確到奈米等級。SPDM能達到每秒16楨與有效的2D解像度精確到10 nm ;大概每2000楨能與SMI資料結合(所需時間大約10秒)而產生高解像度的三維影像(有效的3D光學解像度大概在40-50 nm左右)。而使用快速照相機有些甚至能有較快的速率(達到每秒數百楨)。而使用合適的染劑能產生較好的3D解析影像。[10]
應用SPDMphymod結合SMI (兩者都在1996年由克里斯托夫克勒梅的實驗室所發明) 利用3D雙重顏色重建Her2/neu與 Her3的排列模式,這些蛋白團能被定位精確到25 nm。[11]
工業上使用超解析顯微鏡
[編輯]超解析顯微技術既能使用在生醫實驗室還能應用在製藥研究上,在辨識與判斷目標物的價值上特別有用,例如: 生物分子裝置(BBM biomolecular machines) 都是由許多勝任基本功能的體細胞大分子所組成高度奈米結構的複合物,依功能而決定特定的結構,BBM的其中一個例子是擬核區,它讓兩公尺長的DNA折疊在直徑只有幾微米的區域,因此DNA 才能在細胞中扮演訊息與調控的中心腳色。
藉由使用LIMON 3D結合LIMON複合物標定,使第一次做3D複合物如BMM的隱密蛋白質或核酸能顯像且不破壞其結構。直到現今,最大的問題是在詳細分析內部大分子時必須破壞複合物的構造,所以有些利用電腦模型或者以核磁共振方式來模擬複合物的三維結構。[12]
相關文獻
[編輯]- ^ Reymann, J; Baddeley, D; Gunkel, M; Lemmer, P; Stadter, W; Jegou, T; Rippe, K; Cremer, C; Birk, U. High-precision structural analysis of subnuclear complexes in fixed and live cells via spatially modulated illumination (SMI) microscopy. Chromosome research : an international journal on the molecular, supramolecular and evolutionary aspects of chromosome biology. 2008, 16 (3): 367–82. PMID 18461478. doi:10.1007/s10577-008-1238-2.
- ^ R. Heintzmann, C. Cremer (1999) Lateral modulated excitation microscopy: Improvement of resolution by using a diffraction grating. Proc. SPIE 3568: 185-196
- ^ US patent 7,342,717: Christoph Cremer, Michael Hausmann, Joachim Bradl, Bernhard Schneider Wave field microscope with detection point spread function, priority date 10th july 1997
- ^ Best G, Amberger R, Baddeley D, Ach T, Dithmar S, Heintzmann R and Cremer C (2011). Structured illumination microscopy of autofluorescent aggregations in human tissue. Micron, 42, 330-335
- ^ Lemmer P, Gunkel M, Baddeley D, Kaufmann R, Urich A, Weiland Y, Reymann J, Müller P, Hausmann M, Cremer C (2007): SPDM – Light Microscopy with Single Molecule Resolution at the Nanoscale. In: Applied Physics B 2008; 93: 1–12
- ^ J. Bradl, B. Rinke, A. Esa, P. Edelmann, H. Krieger, B. Schneider, M. Hausmann, C. Cremer (1996): Comparative study of three-dimensional localization accuracy in conventional, confocal laser scanning and axialtomographic fluorescence light microscopy. Proc. SPIE 2926: 201-206
- ^ R. Heintzmann, H. Münch, C. Cremer (1997) High-precision measurements in epifluorescent microscopy - simulation and experiment. Cell Vision 4: 252-253
- ^ US patent 6,424,421: Christoph Cremer, Michael Hausmann, Joachim Bradl, Bernd Rinke Method and devices for measuring distances between object structures, priority date 23rd december 1996
- ^ Manuel Gunkel, Fabian Erdel, Karsten Rippe, Paul Lemmer, Rainer Kaufmann, Christoph Hörmann, Roman Amberger and Christoph Cremer (2009): Dual color localization microscopy of cellular nanostructures. In: Biotechnology Journal, 2009, 4, 927-938. ISSN 1860-6768
- ^ Baddeley D, Batram C, Weiland Y, Cremer C, Birk UJ.: Nanostructure analysis using Spatially Modulated Illumination microscopy. In: Nature Protocols 2007; 2: 2640–2646
- ^ Rainer Kaufmann, Patrick Müller, Georg Hildenbrand, Michael Hausmann & Christoph Cremer (2010): Analysis of Her2/neu membrane protein clusters in different types of breast cancer cells using localization microscopy, Journal of Microscopy 2010, doi:10.1111/j.1365-2818.2010.03436.x
- ^ Super Resolution Microscopy for Pharmaceutical Industry: Patents granted for 3D complex labeling. [2013-07-01]. (原始內容存檔於2012-01-09).
相關連結
[編輯]- GFP Superresolution
- Publication list optical nanoscopy(頁面存檔備份,存於互聯網檔案館)
- press release of the University of Heidelberg(頁面存檔備份,存於互聯網檔案館)
- Superresolution Measurements Laser Optical Nanoscopy
| |||||||||||||||||||||||||

