DNA納米技術
DNA納米技術專門研究利用脫氧核糖核酸或其他核酸的分子性質(如自組裝的特性),來建構出可操控的新型納米尺度結構或機械。在這個領域,核酸被用作非生物的材料而不是在活細胞中那樣作為遺傳信息的載體。嚴格的核酸鹼基配對法則(使鏈上特定的鹼基列相互連接以形成牢固的雙螺旋結構)使這一技術成為可能。這一技術允許合理的鹼基鏈設計,從而嚴格地組合形成具有精密控制的納米級特性的複雜的目標結構。脫氧核糖核酸是常使用的優勢材料,但包括其他核酸如核糖核酸和肽核酸也被用來構造結構,所以偶爾也用「核酸納米技術」來概括這個領域。[2][3]
DNA納米技術概念的基礎最先由納德里安·西曼(Nadrian Seeman)在1980年代早期闡述,在2000年後開始引起廣泛的關注。這一領域的研究者已經構建了靜止結構如二維和三維晶體結構、毫微管、多面體和其他任意的造型;和功能結構如納米機器和DNA運算。一些組建方法被用來構建拼裝結構、摺疊結構和動態可重構結構。現在,這種科技開始被用作解決在結構生物學和生物物理學中基礎科學問題的工具;同時也被應用在結晶學和光譜學中來測定蛋白質結構。這項技術在分子電子學(molecular scale electronics)和納米醫學中的應用仍在研究中。
歷史
[編輯]DNA納米技術概念的基礎最先由納德里安·西曼(Nadrian Seeman) 在八十年代早期闡述。[4]西曼的最初目的是創建一個三維的DNA晶格為其他大分子確定方向,這將去除獲得純淨晶體的複雜過程,簡化晶體學研究。據說他在意識到莫里茨·科內利斯·埃舍爾(M. C. Escher)的木刻Depth與大批的六節點DNA間的共同點後,產生了這個想法。[5][6]在當時,一些自然地有分支的DNA結構已被人們知曉包括複製叉和可移動的霍利迪交叉,但西曼認為不可動的核酸節點可以通過正確的鹼基序列設計消除組合分子的對稱性來製造。那樣這些不可動節點在原則上可以組合成穩固的晶格。第一篇闡明這個體系的論文於1982年發表,第一份試驗證明於次年刊出。[7][8]
在1991年,西曼的實驗室發布了一份對用DNA製成的立方體(第一個人工合成的三維核酸納米結構)的分析報告。他因此獲得1995年費曼納米技術獎(Feynman Prize in Nanotechnology)。DNA截面八角體也隨之誕生。然而,人們很快發現這些以可變節點作為頂點的多邊形結構對構造伸展的三維晶格來說不夠牢固。西曼發展了更為牢固的雙交叉形(DX),在1998年與Erik Winfree的合作中發布了用DX拼接成的二維晶格。[5][4][9]這些拼接結構具有實施DNA計算(DNA computing)的能力。這一點已由Winfree和Paul Rothemund在他們2004年關於謝爾賓斯基三角形結構(Sierpinski gasket)的論文中證明,他們因此分享了2006年費曼納米技術獎。Winfree的主要觀點是DX磚可以當做王氏磚(Wang tiles)使用,也就是說他們的組合可進行計算。[4]對三維晶格的分析最終由西曼在2009年發表,為完成它西曼花了大約30年。[10]
在2000年代設計的DNA結構的新功能陸續被發現。第一個DNA納米機器—會對輸入做出反應而改變其形態-在1999年由西曼驗證。次年,Bernard Yurke驗證了一個有提升的核酸機器。更進一步的,這被翻譯成機械運動,並在2004和2005年一些DNA行走器件被西曼、Niles Pierce、 Andrew Turberfield和Chengde Mao所驗證。[11]用DNA組列構建其他分子的模板(如納米粒子、蛋白質等)的構想最早由Bruche Robinson 和西曼在1987年提出,在2006、2007年被Hao Yan、Peter Dervan和Thomas Labean驗證。[12][8][13]
在2006年,Rothemund第一次驗證了DNA摺紙技術可以簡單地構建出穩固的具有任意造型的結構。Rothemund設想這種用多條短鏈的方法是介於西曼晶格(用多條短鏈)和William Shih的DNA八面體(主要用一條長鏈)的概念性中產物。Rothemund的DNA摺紙法是用一條長鏈和許多短鏈配合摺疊。這種方法使構建更大的結構變得可行並降低了對設計和分析的技術要求。[9]DNA摺紙術在2006年3月15日登上了《自然》雜誌的封面。[14]隨着Rothemund完成驗二維DNA摺紙結構,Douglas等人在2009年固性三維DNA摺紙術。[15] 同時,Jørgen Kjems的實驗室用二維平面製成了三維結構。[10]
DNA納米技術最初受到一些質疑,因為核酸被當作非生物的材料建造結構和進行計算,並且遠離實際應用。西曼1991年關於DNA立方體分析的論文被科學雜誌拒絕,在此之前,一個評論家稱讚他的獨創性而另一個批評論文與生物學相關性不大。然而,2010年代,這個領域在基礎科學方面研究的應用開始被認識到,並且其在醫學和其他領域的應用被認為是可行的。[10][16]2001年還只有很少的實驗室做這方面研究,到2010年至少有60個實驗室,也提高了人才儲備量。因此在這十年內這方面的研究取得了很大進步。[17]
基礎概念
[編輯]

核酸的特性
[編輯]納米技術常被限定為在小於100納米的尺度下對材料和物體的研究。DNA納米技術是自下而上分子自組裝的一個例子,即分子成分自發地組成穩定結構;這些結構的特殊形態是由設計者所挑選的成分的物化特性所引發的。[19]在DNA納米技術中,構建成分是核酸鏈,如脫氧核糖核酸。脫氧核糖核酸很適合納米級的構造,因為一條脫氧核糖核酸雙螺旋直徑為2nm,螺旋重複長度為3.5nm。核酸比其他材料更適合構造結構的主要特性是兩條核酸鏈間簡單的鹼基配對法則,通過鹼基連接可以形成一個具體明確的結構。這使核酸結構的組合容易通過核酸設計得到控制。這種特性在其他被用於納米技術的材料中並不存在(包括蛋白質和納米顆粒)。[8]
核酸分子的結構由含不同鹼基的核苷酸的排列順序決定。在脫氧核糖核酸中,四個鹼基為腺嘌呤、胞嘧啶、鳥嘌呤和胸腺嘧啶。如果核酸的兩條鏈是互補的,它們將相互連接形成雙螺旋結構(A-T,C-G)。[8][20]因為正確的鹼基配對是積極有利的,在大多數情況下,核酸鏈相互連接時,都要使正確配對的鹼基數目最大化。鏈中鹼基的排列決定了連接的方式和整體結構,這簡單可控。在DNA納米技術中,研究者設計合理的鹼基序列,從而使得整條鏈按照研究者希望的那樣組合起來。[5][8]
分支
[編輯]DNA納米技術有時被分為兩個有所重疊的領域:結構DNA納米技術(structural DNA nanotechnology)和動態DNA納米技術(dynamic DNA nanotechnology)。結構DNA納米技術(有時縮略為SDN)主要方向是合成核酸材料等合成物並組合成穩定平衡的最終形態。[21][22]
而動態DNA納米技術的主要方向是研究具有不平衡性狀(如在物化條件的誘導下具有重組能力)的合成物。一些合成物如核酸納米機械(nucleic acid nanomechanical devices)同時具有這兩個分支的特性。在結構DNA納米技術中構建的合成物會有拓撲分支的核酸結構。(大多數生物DNA是不含分支的雙螺旋結構。)一個最簡單的分支結構是在一個節點上產生四條分支,這包含四股獨立的DNA鏈。不像自然的霍利迪交叉,在這個人工節點上每條分支都有不同的鹼基序列,因此這個節點被固定在某個位置。多個節點可以在同一個合成物中被組合起來,如廣泛使用的雙交叉磚(double-crossover (DX)motif),它由兩條平行的雙螺旋鏈和獨立的在其間交叉的鏈組成。其交叉於兩個點,每個點都是一個四支節點,但是與一個可變的四支節點不同,它被約束到單一的方向,使DX磚作為對更大的DNA合成物的結構障礙。[8][5]
動態DNA納米技術運用了一種叫做toehold-mediated strand displacement的機制使核酸合成物對新核酸鏈的添加做出反應進行重組。在這個反應中,移入的鏈與雙鏈合成物中一條鏈上的點位進行連接,並在分支遷移過程中置換掉初始合成物中的一條鏈。也就是合成物中的一條鏈被另外的鏈所替換。[21]重組結構與機械也可用功能核酸製造,如脫氧核酶、核酶或核酸適體。[23]
設計
[編輯]DNA的納米結構必須被合理設計,單條核酸鏈才會被組合成目標結構。這個過程通常需要對目標結構或功能的詳述。然後確定目標合成物的二級結構即對結構內核酸鏈的安置和連接點位。最後是一級結構的設計即對每條核酸鏈鹼基順序的安排。[24][25]
結構設計
[編輯]設計核酸納米結構的第一步是決定如何用核酸鏈構建目標結構。這一步決定了其二級結構和連接點位。[24]有如下一些方法:
- 拼接結構:這種方法打破部分化學鍵將目標結構分解成小單元。這常被用來做點陣,但也被用來實施系統的自組,使之成為DNA計算的平台。這是從90年代中期DNA摺紙法發展以來十多年的主導設計策略。[24][26]
- 摺疊結構:摺疊法是用一條長鏈構造納米結構。這條長鏈可以有設計好的鹼基序列而自身摺疊或者用短一些的鏈「訂」在一起,摺疊成目標結構。這後一種方法叫做DNA摺紙(DNA origami)。通過這種方法,構建納米級的二維或三維形態也得以實現(見下面的非連續結構)。[7][14]
- 動態組合:這種方法直接控制DNA自組的動力,除了中產物外要分析反應中所有的中間步驟。用採用了髮夾結構的原始材料;這些發生級聯反應以一個特定的順序形成最終構造。這種方法有在恆定溫度下進行的優勢。這不同於熱力學方法,它不需要熱退火步驟來使之形成目標結構。[7][27]
序列設計
[編輯]在用上述方法設計了目標合成物的二級結構後,核苷酸的序列必須要確定出。許多方法都以目標結構具有最低能量為目的設計序列,因為這在熱力學上是最有利的,而拆卸的結構具有更高的能量,因此不有利。這樣做是可以通過更快的、簡單的、啟發性的方法如序列對稱最小化,或使用一個完整的最鄰近(nearest-neighbor)熱力學模型,這更精確但更慢並且需要更密集的計算。幾何模型用來測定納米結構的三級結構以保證合成物不會過度緊密。[25][28]
核酸設計與蛋白質設計有相似的目標。在兩者中,單體的序列被設計成有利於目標結構而不利於其他結構。核酸設計比蛋白質設計的計算要更簡單,因為簡單的鹼基配對法則足夠預測結構的能量支持(energetic favorability),並且關於總體上結構的三維摺疊的細節信息是不需要的。這允許使用簡單的啟發式方法產生穩固的實驗性設計。然而,核酸結構在功能上多樣性比蛋白質弱,因為有更強的摺疊成合成結構,並且與20種氨基酸相比,四種鹼基缺少化學多樣性。[28]
結構DNA納米技術
[編輯]
結構DNA納米技術(有時縮寫為SDN),着重於對核酸合成物的分析與描述。核酸的雙螺旋是一個穩固的三維幾何結構,這使得預測和設計更為複雜的核酸合成物成為可能。許多這樣的結構已經被創造出來包括二維、三維結構,周期性的、非周期性的和離散結構。[22]
擴展晶格
[編輯]小的核酸合成物可以配上粘性末端,合成更大的二維周期性晶格(含由個體磚組建的特殊的密鋪型)。[22]最早的一個例子是用DX磚作為基礎磚,每個磚都包含四個設計好的粘性末端,以使DX磚可以合成二維平面結構。這基本上是剛性的DNA二維晶格。[32][33]二維組列也可以用其他形製造如霍利迪交叉菱形晶格和各種以DX磚為基礎的組列。[34][35]上面兩幅圖便是周期性晶格的例子。
二維組列可以表現出非周期性結構,它們的組合能夠執行運算,展示出DNA計算的一種形式。[17]DX磚可以通過選擇帶粘性末端的序列充當王氏磚,並執行運算DX組列。能夠編碼邏輯異或程序的DX組列已被驗證;這使DNA組列可以實施一個細胞自動機,生成一個不規則分形(謝爾賓斯基三角形結構)。右邊第三個圖片展現了這個組列的形式。[31]另一個系統有二進制計數功能,它的增長意味着二進制數的增加。[36]
DX組列已經能被做出直徑4-20nm的中空納米管,這主要是二維晶格自身捲起。[24]這些DNA納米管在形狀大小上和碳納米管有一點相似,但缺少碳納米管的電導。DNA納米管更容易被修改、被連接到其他結構。構建DNA納米碳管的其中一個方案是用一個DX磚晶格,它自身捲起成管。[37]另一種方法是用單鏈磚使圓周被指定在一個簡單的模塊化的形式,這個管的剛性是一個新生特性。[38]
用DNA創建三維晶格是DNA納米技術最早的目標,但這很難實現。最終在2009年,報道稱成功地使用建立在無尺寸限制結構的概念上的圖形創建出三維結構並在緊張壓縮間達到平衡。[17][39]
離散結構
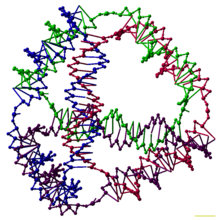
[編輯]研究者已經分析了一些與多面體相關的三維DNA合成物,每個都與多面體有聯通性,比如立方體或八面體,這意味着DNA雙鏈是多面體的棱而每個頂點都是一個DNA節點。[7]早期對DNA多面體的驗證工作量很大,需要複雜的結紮和固相結合的步驟來建造連續的多面體。[40]隨後的工作使多面體的分析變得簡單。[41]這些包括一個用一條長鏈製造DNA八面體,和一步用四條DNA鏈製成一個四面體。(見上圖)[1]
任意的納米結構、不規則的形狀通常用DNA摺紙法製成。這些結構有一條病毒鏈作為支架,這被用計算設計的短鏈訂成目標形狀。這種方法有容易設計的優勢,因為鹼基序列是被支架鹼基序列所預先確定的,並且不需要高的鏈純度和精確的化學計量。DNA摺紙法首先在二維形中使用,比如表情符號和粗略的北美地圖。[7][14]剛性的三維結構可以通過使用安排成蜂窩狀的DNA雙螺旋製造;[15]二維平面可以被摺疊成中空的三維形狀,類似於一個箱子。這些可以被編碼為對於刺激做出反應,使它們作為分子籠。[42][43]
模板組裝
[編輯]核酸分子可以吸收其他分子如蛋白質金屬納米離子、量子點(quantum dots)、富勒烯。這使得構建具有多種功能的機械變得易行。用核酸結構的自組來作為附加粒子自組的樣板,控制它們的位置,並在一些情況下定位。[7][13]許多方案使用共價結合的辦法,用酰胺或硫醇為官能團的寡核酸鏈綁定異質粒子。[44]這種共價結合的辦法已經被用來把金納米粒子和鏈霉親和素分子固定在DX磚組列上。[45]有一種非共價結合法用Dervan聚酰胺。[46]碳納米管以某種形狀被連接到DNA組列上充當分子電子機器——碳納米管場效應晶體管。[47]另外,還有核酸金屬化法即用金屬代替核酸呈現最初的核酸結構。——用核酸納米結構作為模具,將形狀轉化為固體型。[48][49]
動態DNA納米技術
[編輯]
動態DNA納米技術着重於建造具有動態功能(如計算和機械運動)的與整體結構相關的核酸系統。結構DNA納米技術和動態DNA納米技術有所重疊。因為結構可以通過退火和動態重構形成或者首先動態組合。[7][11]
納米機械裝置
[編輯]人們已經製造出隨着一些外界刺激改變造型的DNA合成物——這是納米機器人的一種形式。這些結構最初都是以與構建靜止結構相同的方式形成的;但經過設計,於是在最初的形成後能夠動態重構。[21][11]最早的這樣的機械利用了B-DNA與Z-DNA間的轉變來對在緩衝環境下通過扭轉運動做出反應。[50]然而,這倚賴於緩衝液的條件使所有的機械在同一時間改變狀態。後續的系統可以根據控制鏈的存在改變狀態,使多種機械可在溶液中獨立地運轉。這樣系統的一個例子是「分子鑷」設計。[51]它有開和閉的形式,在旋轉運動過程中從平行交叉構造(paranemic-crossover (PX))轉為雙節構造(double-junction (JX2))。[52]另有一種二維組列可以動態地響應控制連進行擴張或收縮。[53]人們已造出可以動態開關,能作為「分子籠」在打開時釋放功能「貨物」。[42][54][55]
DNA行走者(DNA walkers)是一類沿直線軌道運動的核酸納米機器。大量的方案已被驗證。[11]其中一個方案是用需人工添加序列的控制鏈控制行走者沿軌跡的移動。[56][57]另一個方案是利用限制酶或脫氧核酶解開鏈,使行走者前移,這種方法的優點是自動運行。[58][59]後來一種系統可以在二維平面而不是一條直線上運動,並且被證實有選擇性地獲取和移動分子貨物的功能。[60]另外,一個直線行走者被證實在其移動的過程中會進行DNA合成。[61]
鏈置換級聯反應
[編輯]不論計算或結構目的鏈置換級聯反應都可以使用。單獨的鏈鏈置換反應包括對一些引發鏈的存在做出反應產生新序列。許多這樣的反應被連到級聯上,一個反應新產生的序列可以引起另一個鏈置換級聯反應。這反過來又促進了化學反應網絡的建設,表現出複雜的計算和信息處理能力。通過新鍵的形成和從分解反應中獲得的熵,級聯變得積極有利。鏈置換級聯反應允許組合與計算過程中的等溫操作(這與傳統核酸組合要求熱退火工序相反)。它們也支持催化功能,小於一當量的引發劑就可以使反應完成。[21][27]
鏈置換合成物可以用來製造能執行複雜運算的分子邏輯門。不像用電流作為輸入和輸出的傳統的電子計算機,分子計算機用特定化學物質的濃度作為信號。在核酸鏈置換電路中,信號是核酸與其他鏈的連接與斷開釋放或吸收的核酸。這種方法已被用來做與、或、非門。[62]最近,一個四位電路被驗證可以做0-15整數平方根的計算(用包含130條DNA的邏輯門)。[63]
鏈置換級聯反應的另一個作用是製造動態組合結構。這些用髮夾結構作為反應物,當輸入的鏈連接時,新序列會在同一分子上而不是分解。這使新打開的髮夾添加到合成物中。這種方法已被用來做簡單的結構如三或四支節點和樹狀物。[27]
應用
[編輯]DNA納米技術提供了設計構建複雜結構並精確控制納米特性的一種方法。這個領域開始應用在結構生物學與生物物理學中解決基礎科學問題。最初,DNA納米技術設想應用於晶體學研究上:難以在隔離狀態下結晶的分子可被置於立體核酸晶格中從而測定其的結構。另一種應用是在蛋白質核磁共振波譜學中的殘基偶極耦合(RDC)實驗中用DNA摺紙棒來代替液晶。用DNA摺紙術是非常有好處的,因為不像液晶,DNA摺紙棒可以承受使膜蛋白懸浮的洗滌劑。DNA 行走者(DNA walkers)被用作納米組合線來移動納米粒子,指導化學合成。不僅如此,DNA摺紙結構也促進了酶功能和蛋白質摺疊方面的研究。[22][10]
DNA納米技術正朝潛在的實際應用邁步。核酸組列安置其他分子的能力暗示了其在分子電子學上的應用前景。核酸結構可以被用作分子電子單位(如分子導線(molecular wires))組合的模板,提供對安置進行控制的方法,類似於一個電路實驗板。[22][7]DNA納米技術被比作可程式化材料,因為其材料的耦合計算。[64]
DNA納米技術在納米機械方面也有潛在的應用。比如利用其以生物相容性的計算製藥,使之能夠靶向給藥(targeted drug delivery)。一個現在正被研究的系統用了一個中空的DNA盒子,其中包着可以引發細胞凋亡或死亡的蛋白質,而只有靠近癌細胞時,盒子才會打開,釋放出蛋白質。[10][65]人們對在細胞中表達人工結構懷着極大興趣,最有可能使用轉錄RNA進行組裝,儘管仍不知道這些合成結構是否能有效地摺疊或聚集在細胞質中。[7]
材料與方法
[編輯]
DNA鏈是用分子建模和熱力學建模軟件計算設計的。[25][28]核酸用標準的寡核苷酸合成法合成,通常用寡核苷酸合成器自動進行合成,自定義序列的鏈是市售的。[66]如果需要的話,鏈可以通過變性凝膠電泳純化,精確的濃度可用紫外線吸收光譜來定量測定。[67][68]
完全形成的目標結構可用非變性凝膠電泳檢驗,將會得到關於核酸合成物形態大小的信息。電泳遷移率變動分析可以評估一個結構是否包含所有的所需鏈。[69]熒光標記和熒光共振能量轉移(FRET)有時用一表示合成物結構的特性。[70]
核酸結構可以直接用原子力顯微鏡看到,這很適合延伸的二維結構,但對三維結構沒有多大用處,因為顯微鏡尖端會與脆弱的核酸結構相互作用;因此透射電子顯微鏡和低溫電子顯微鏡常用以觀察三維結構。延伸的三維晶格用X光晶體學分析。[71][72]
參見
[編輯]參考文獻
[編輯]- ^ 1.0 1.1 DNA polyhedra: Goodman, Russel P.; Schaap, Iwan A. T.; Tardin, C. F.; Erben, Christof M.; Berry, Richard M.; Schmidt, C.F.; Turberfield, Andrew J. Rapid chiral assembly of rigid DNA building blocks for molecular nanofabrication. Science. 9 December 2005, 310 (5754): 1661–1665. Bibcode:2005Sci...310.1661G. PMID 16339440. doi:10.1126/science.1120367. (英文)
- ^ RNA nanotechnology: Chworos, Arkadiusz; Severcan, Isil; Koyfman, Alexey Y.; Weinkam, Patrick; Oroudjev, Emin; Hansma, Helen G.; Jaeger, Luc. Building Programmable Jigsaw Puzzles with RNA. Science. 2004, 306 (5704): 2068–2072. Bibcode:2004Sci...306.2068C. PMID 15604402. doi:10.1126/science.1104686.(英文)
- ^ RNA nanotechnology: Guo, Peixuan. The Emerging Field of RNA Nanotechnology. Nature Nanotechnology. 2010, 5 (12): 833–842. Bibcode:2010NatNa...5..833G. PMC 3149862
 . PMID 21102465. doi:10.1038/nnano.2010.231.(英文)
. PMID 21102465. doi:10.1038/nnano.2010.231.(英文)
- ^ 4.0 4.1 4.2 History: Pelesko, John A. Self-assembly: the science of things that put themselves together. New York: Chapman & Hall/CRC. 2007: 201, 242, 259. ISBN 978-1-58488-687-7.(英文)
- ^ 5.0 5.1 5.2 5.3 5.4 Overview: Seeman, Nadrian C. Nanotechnology and the double helix. Scientific American. June 2004, 290 (6): 64–75 [2012-08-28]. PMID 15195395. doi:10.1038/scientificamerican0604-64. (原始內容存檔於2013-11-10).(英文)
- ^ History: See Current crystallization protocol. Nadrian Seeman Lab. [2012-08-28]. (原始內容存檔於2012-11-30). for a statement of the problem, and DNA cages containing oriented guests. Nadiran Seeman Laboratory. [2012-08-28]. (原始內容存檔於2016-11-01). for the proposed solution.(英文)
- ^ 7.0 7.1 7.2 7.3 7.4 7.5 7.6 7.7 7.8 Overview: Andre V. Pinheiro, Dongran Han, William M. Shih, Hao Yan. Challenges and opportunities for structural DNA nanotechnology. Nature Nanotechnology. 2011/12, 6 (12): 763–772 [2018-04-02]. ISSN 1748-3395. doi:10.1038/nnano.2011.187. (原始內容存檔於2019-02-15) (英語).
- ^ 8.0 8.1 8.2 8.3 8.4 8.5 Overview: Nadrian C. Seeman. Nanomaterials Based on DNA. Annual Review of Biochemistry. 2010-06-07, 79 (1): 65–87 [2018-04-02]. ISSN 0066-4154. doi:10.1146/annurev-biochem-060308-102244. (原始內容存檔於2019-02-15).(英文)
- ^ 9.0 9.1 DNA origami: Rothemund, Paul W. K. Scaffolded DNA origami: from generalized multicrossovers to polygonal networks. Chen, Junghuei; Jonoska, Natasha; Rozenberg, Grzegorz (編). Nanotechnology: science and computation. Natural Computing Series. New York: Springer. 2006: 3–21. ISBN 978-3-540-30295-7. doi:10.1007/3-540-30296-4_1.(英文)
- ^ 10.0 10.1 10.2 10.3 10.4 History/applications: Service, Robert F. DNA nanotechnology grows up. Science. 3 June 2011, 332 (6034): 1140–1143. doi:10.1126/science.332.6034.1140.(英文)
- ^ 11.0 11.1 11.2 11.3 DNA machines: Bath, Jonathan; Turberfield, Andrew J. DNA nanomachines. Nature Nanotechnology. May 2007, 2 (5): 275–284. Bibcode:2007NatNa...2..275B. PMID 18654284. doi:10.1038/nnano.2007.104.(英文)
- ^ Nanoarchitecture: Robinson, Bruche H.; Seeman, Nadrian C. The design of a biochip: a self-assembling molecular-scale memory device. Protein Engineering. August 1987, 1 (4): 295–300. PMID 3508280. doi:10.1093/protein/1.4.295. (英文)
- ^ 13.0 13.1 Overview: Masayuki Endo, Hiroshi Sugiyama. Chemical Approaches to DNA Nanotechnology. ChemBioChem. 2009-10-12, 10 (15): 2420–2443 [2018-04-02]. ISSN 1439-7633. doi:10.1002/cbic.200900286 (英語).
- ^ 14.0 14.1 14.2 DNA origami: Rothemund, Paul W. K. Folding DNA to create nanoscale shapes and patterns. Nature. 16 March 2006, 440 (7082): 297–302. Bibcode:2006Natur.440..297R. PMID 16541064. doi:10.1038/nature04586.(英文)
- ^ 15.0 15.1 DNA origami: Douglas, Shawn M.; Dietz, Hendrik; Liedl, Tim; Högberg, Björn; Graf, Franziska; Shih, William M. Self-assembly of DNA into nanoscale three-dimensional shapes. Nature. 21 May 2009, 459 (7245): 414–418. Bibcode:2009Natur.459..414D. PMC 2688462
 . PMID 19458720. doi:10.1038/nature08016.(英文)
. PMID 19458720. doi:10.1038/nature08016.(英文)
- ^ History: Hopkin, Karen. Profile: 3-D seer. The Scientist. August 2011 [8 August 2011]. (原始內容存檔於2011-10-10).(英文)
- ^ 17.0 17.1 17.2 History: Seeman, Nadrian. Structural DNA nanotechnology: growing along with Nano Letters. Nano Letters. 9 June 2010, 10 (6): 1971–1978. Bibcode:2010NanoL..10.1971S. PMC 2901229
 . PMID 20486672. doi:10.1021/nl101262u.(英文)
. PMID 20486672. doi:10.1021/nl101262u.(英文)
- ^ 18.0 18.1 18.2 Overview: Mao, Chengde. The emergence of complexity: lessons from DNA. PLoS Biology. December 2004, 2 (12): 2036–2038. PMC 535573
 . PMID 15597116. doi:10.1371/journal.pbio.0020431.(英文)
. PMID 15597116. doi:10.1371/journal.pbio.0020431.(英文)
- ^ Background: Pelesko, John A. Self-assembly: the science of things that put themselves together. New York: Chapman & Hall/CRC. 2007: 5, 7. ISBN 978-1-58488-687-7.(英文)
- ^ Background: Long, Eric C. Fundamentals of nucleic acids. Hecht, Sidney M (編). Bioorganic chemistry: nucleic acids. New York: Oxford University Press. 1996: 4–10. ISBN 0-19-508467-5.(英文)
- ^ 21.0 21.1 21.2 21.3 Dynamic DNA nanotechnology: David Yu Zhang, Georg Seelig. Dynamic DNA nanotechnology using strand-displacement reactions. Nature Chemistry. 2011/02, 3 (2): 103–113 [2018-04-02]. ISSN 1755-4349. doi:10.1038/nchem.957. (原始內容存檔於2019-09-25) (英語).
- ^ 22.0 22.1 22.2 22.3 22.4 Structural DNA nanotechnology: Seeman, Nadrian C. An overview of structural DNA nanotechnology. Molecular Biotechnology. November 2007, 37 (3): 246–257. PMID 17952671. doi:10.1007/s12033-007-0059-4.(英文)
- ^ Dynamic DNA nanotechnology: Yi Lu, Juewen Liu. Functional DNA nanotechnology: emerging applications of DNAzymes and aptamers. Current Opinion in Biotechnology: 580–588. [2018-04-02]. doi:10.1016/j.copbio.2006.10.004. (原始內容存檔於2018-06-12).(英文)
- ^ 24.0 24.1 24.2 24.3 Design: Udo Feldkamp, Christof M. Niemeyer. Rational Design of DNA Nanoarchitectures. Angewandte Chemie International Edition. 2006-03-13, 45 (12): 1856–1876 [2018-04-02]. ISSN 1521-3773. doi:10.1002/anie.200502358 (英語).
- ^ 25.0 25.1 25.2 Design: Brenneman, Arwen; Condon, Anne. Strand design for biomolecular computation. Theoretical Computer Science. 25 September 2002, 287: 39–58. doi:10.1016/S0304-3975(02)00135-4.(英文)
- ^ Overview: Lin, Chenxiang; Liu, Yan; Rinker, Sherri; Yan, Hao. DNA tile based self-assembly: building complex nanoarchitectures. ChemPhysChem. 11 August 2006, 7 (8): 1641–1647. PMID 16832805. doi:10.1002/cphc.200600260.(英文)
- ^ 27.0 27.1 27.2 Kinetic assembly: Yin, Peng; Choi, Harry M. T.; Calvert, Colby R.; Pierce, Niles A. Programming biomolecular self-assembly pathways. Nature. 17 January 2008, 451 (7176): 318–322. Bibcode:2008Natur.451..318Y. PMID 18202654. doi:10.1038/nature06451.(英文)
- ^ 28.0 28.1 28.2 Design: Dirks, Robert M.; Lin, Milo; Winfree, Erik & Pierce, Niles A. Paradigms for computational nucleic acid design. Nucleic Acids Research. 15 February 2004, 32 (4): 1392–1403. PMC 390280
 . PMID 14990744. doi:10.1093/nar/gkh291. (英文)
. PMID 14990744. doi:10.1093/nar/gkh291. (英文)
- ^ Other arrays: Strong, Michael. Protein Nanomachines. PLoS Biology. March 2004, 2 (3): e73. PMC 368168
 . PMID 15024422. doi:10.1371/journal.pbio.0020073.
. PMID 15024422. doi:10.1371/journal.pbio.0020073.
- ^ Hao Yan, Sung Ha Park, Gleb Finkelstein, John H. Reif, Thomas H. LaBean. DNA-Templated Self-Assembly of Protein Arrays and Highly Conductive Nanowires. Science. 2003-09-26, 301 (5641): 1882–1884 [2018-04-02]. ISSN 0036-8075. doi:10.1126/science.1089389. (原始內容存檔於2018-04-30) (英語).
- ^ 31.0 31.1 Algorithmic self-assembly: Rothemund, Paul W. K.; Papadakis, Nick; Winfree, Erik. Algorithmic self-assembly of DNA Sierpinski triangles. PLoS Biology. December 2004, 2 (12): 2041–2053. PMC 534809
 . PMID 15583715. doi:10.1371/journal.pbio.0020424. (英文)
. PMID 15583715. doi:10.1371/journal.pbio.0020424. (英文)
- ^ DX arrays: Winfree, Erik; Liu, Furong; Wenzler, Lisa A.; Seeman, Nadrian C. Design and self-assembly of two-dimensional DNA crystals. Nature. 6 August 1998, 394 (6693): 529–544. Bibcode:1998Natur.394..539W. PMID 9707114. doi:10.1038/28998. (英文)
- ^ DX arrays: Liu, Furong; Sha, Ruojie; Seeman, Nadrian C. Modifying the surface features of two-dimensional DNA crystals. Journal of the American Chemical Society. 10 February 1999, 121 (5): 917–922. doi:10.1021/ja982824a. (英文)
- ^ Other arrays: Constantinou, Pamela E.; Wang, Tong; Kopatsch, Jens; Israel, Lisa B.; Zhang, Xiaoping; Ding, Baoquan; Sherman, William B.; Wang, Xing; Zheng, Jianping; Sha, Ruojie; Seeman, Nadrian C. Double cohesion in structural DNA nanotechnology. Organic and Biomolecular Chemistry. 21 September 2006, 4 (18): 3414–3419. PMID 17036134. doi:10.1039/b605212f. (英文)
- ^ Other arrays: Mathieu, Frederick; Liao, Shiping; Kopatsch, Jens; Wang, Tong; Mao, Chengde; Seeman, Nadrian C. Six-helix bundles designed from DNA. Nano Letters. April 2005, 5 (4): 661–665. Bibcode:2005NanoL...5..661M. PMID 15826105. doi:10.1021/nl050084f. (英文)
- ^ Algorithmic self-assembly: Barish, Robert D.; Rothemund, Paul W. K.; Winfree, Erik. Two computational primitives for algorithmic self-assembly: copying and counting. Nano Letters. December 2005, 5 (12): 2586–2592. Bibcode:2005NanoL...5.2586B. PMID 16351220. doi:10.1021/nl052038l.(英文)
- ^ DNA nanotubes: Rothemund, Paul W. K.; Ekani-Nkodo, Axel; Papadakis, Nick; Kumar, Ashish; Fygenson, Deborah Kuchnir & Winfree, Erik. Design and Characterization of Programmable DNA Nanotubes. Journal of the American Chemical Society. 22 December 2004, 126 (50): 16344–16352. PMID 15600335. doi:10.1021/ja044319l. (英文)
- ^ DNA nanotubes: Yin, P.; Hariadi, R. F.; Sahu, S.; Choi, H. M. T.; Park, S. H.; Labean, T. H.; Reif, J. H. Programming DNA Tube Circumferences. Science. 8 August 2008, 321 (5890): 824–826. Bibcode:2008Sci...321..824Y. PMID 18687961. doi:10.1126/science.1157312.(英文)
- ^ Three-dimensional arrays: Zheng, Jianping; Birktoft, Jens J.; Chen, Yi; Wang, Tong; Sha, Ruojie; Constantinou, Pamela E.; Ginell, Stephan L.; Mao, Chengde; Seeman, Nadrian C. From molecular to macroscopic via the rational design of a self-assembled 3D DNA crystal. Nature. 3 September 2009, 461 (7260): 74–77. Bibcode:2009Natur.461...74Z. PMC 2764300
 . PMID 19727196. doi:10.1038/nature08274. (英文)
. PMID 19727196. doi:10.1038/nature08274. (英文)
- ^ DNA polyhedra: Zhang, Yuwen; Seeman, Nadrian C. Construction of a DNA-truncated octahedron. Journal of the American Chemical Society. 1 March 1994, 116 (5): 1661–1669. doi:10.1021/ja00084a006.
- ^ DNA polyhedra: Shih, William M.; Quispe, Joel D.; Joyce, Gerald F. A 1.7-kilobase single-stranded DNA that folds into a nanoscale octahedron. Nature. 12 February 2004, 427 (6975): 618–621. PMID 14961116. doi:10.1038/nature02307. (英文)
- ^ 42.0 42.1 DNA boxes: Andersen, Ebbe S.; Dong, Mingdong; Nielsen, Morten M.; Jahn, Kasper; Subramani, Ramesh; Mamdouh, Wael; Golas, Monika M.; Sander, Bjoern; Stark, Holger. Self-assembly of a nanoscale DNA box with a controllable lid. Nature. 7 May 2009, 459 (7243): 73–76. Bibcode:2009Natur.459...73A. PMID 19424153. doi:10.1038/nature07971. (英文)
- ^ DNA boxes: Ke, Yonggang; Sharma, Jaswinder; Liu, Minghui; Jahn, Kasper; Liu, Yan; Yan, Hao. Scaffolded DNA origami of a DNA tetrahedron molecular container. Nano Letters. 10 June 2009, 9 (6): 2445–2447. Bibcode:2009NanoL...9.2445K. PMID 19419184. doi:10.1021/nl901165f.(英文)
- ^ Nanoarchitecture: Zheng, Jiwen; Constantinou, Pamela E.; Micheel, Christine; Alivisatos, A. Paul; Kiehl, Richard A.; Seeman Nadrian C. 2D Nanoparticle Arrays Show the Organizational Power of Robust DNA Motifs. Nano Letters. July 2006, 6 (7): 1502–1504. Bibcode:2006NanoL...6.1502Z. PMID 16834438. doi:10.1021/nl060994c. (英文)
- ^ Nanoarchitecture: Park, Sung Ha; Pistol, Constantin; Ahn, Sang Jung; Reif, John H.; Lebeck, Alvin R.; Dwyer, Chris; LaBean, Thomas H. Finite-size, fully addressable DNA tile lattices formed by hierarchical assembly procedures. Angewandte Chemie. October 2006, 118 (40): 749–753. doi:10.1002/ange.200690141. (英文)
- ^ Nanoarchitecture: Cohen, Justin D.; Sadowski, John P.; Dervan, Peter B. Addressing single molecules on DNA nanostructures. Angewandte Chemie International Edition. 22 October 2007, 46 (42): 7956–7959. PMID 17763481. doi:10.1002/anie.200702767. (英文)
- ^ Nanoarchitecture: Maune, Hareem T.; Han, Si-Ping; Barish, Robert D.; Bockrath, Marc; Goddard, III, William A.; Rothemund, Paul W. K.; Winfree, Erik. Self-assembly of carbon nanotubes into two-dimensional geometries using DNA origami templates. Nature Nanotechnology. January 2009, 5 (1): 61–66. Bibcode:2010NatNa...5...61M. PMID 19898497. doi:10.1038/nnano.2009.311.(英文)
- ^ Nanoarchitecture: Jianfei Liu, Yanli Geng, Elisabeth Pound, Shailendra Gyawali, Jeffrey R. Ashton, John Hickey, Adam T. Woolley, John N. Harb. Metallization of Branched DNA Origami for Nanoelectronic Circuit Fabrication. ACS Nano. 2011-03-22, 5 (3): 2240–2247 [2018-04-02]. ISSN 1936-0851. doi:10.1021/nn1035075.(英文)
- ^ Nanoarchitecture: Zhaoxiang Deng, Chengde Mao. Molecular Lithography with DNA Nanostructures. Angewandte Chemie International Edition. 2004-08-06, 43 (31): 4068–4070 [2018-04-02]. ISSN 1521-3773. doi:10.1002/anie.200460257 (英語).
- ^ DNA machines: Mao, Chengde; Sun, Weiqiong; Shen, Zhiyong; Seeman, Nadrian C. A DNA nanomechanical device based on the B-Z transition. Nature. 14 January 1999, 397 (6715): 144–146. Bibcode:1999Natur.397..144M. PMID 9923675. doi:10.1038/16437.
- ^ DNA machines: Yurke, Bernard; Turberfield, Andrew J.; Mills, Allen P., Jr; Simmel, Friedrich C.; Neumann, Jennifer L. A DNA-fuelled molecular machine made of DNA. Nature. 10 August 2000, 406 (6796): 605–609. Bibcode:2000Natur.406..605Y. PMID 10949296. doi:10.1038/35020524. (英文)
- ^ DNA machines: Yan, Hao; Zhang, Xiaoping; Shen, Zhiyong; Seeman, Nadrian C. A robust DNA mechanical device controlled by hybridization topology. Nature. 3 January 2002, 415 (6867): 62–65. Bibcode:2002Natur.415...62Y. PMID 11780115. doi:10.1038/415062a.
- ^ DNA machines: Liping Feng, Sung Ha Park, John H. Reif, Hao Yan. A Two-State DNA Lattice Switched by DNA Nanoactuator. Angewandte Chemie. 2003-09-22, 115 (36): 4478–4482 [2018-04-02]. ISSN 1521-3757. doi:10.1002/ange.200351818 (英語).
- ^ DNA machines: Russell P. Goodman, Mike Heilemann, Sören Doose, Christoph M. Erben, Achillefs N. Kapanidis, Andrew J. Turberfield. Reconfigurable, braced, three-dimensional DNA nanostructures. Nature Nanotechnology. 2008/02, 3 (2): 93–96 [2018-04-02]. ISSN 1748-3395. doi:10.1038/nnano.2008.3. (原始內容存檔於2019-09-24) (英語).
- ^ Applications: Shawn M. Douglas, Ido Bachelet, George M. Church. A Logic-Gated Nanorobot for Targeted Transport of Molecular Payloads. Science. 2012-02-17, 335 (6070): 831–834 [2018-04-02]. ISSN 0036-8075. doi:10.1126/science.1214081. (原始內容存檔於2018-04-15) (英語).
- ^ DNA walkers: Shin, Jong-Shik; Pierce, Niles A. A synthetic DNA walker for molecular transport. Journal of the American Chemical Society. 8 September 2004, 126 (35): 10834–10835. PMID 15339155. doi:10.1021/ja047543j.(英文)
- ^ DNA walkers: Sherman, William B.; Seeman, Nadrian C. A precisely controlled DNA biped walking device. Nano Letters. July 2004, 4 (7): 1203–1207. Bibcode:2004NanoL...4.1203S. doi:10.1021/nl049527q.(英文)
- ^ DNA walkers: Tian, Ye; He, Yu; Chen, Yi; Yin, Peng; Mao, Chengde. A DNAzyme that walks processively and autonomously along a one-dimensional track. Angewandte Chemie. 11 July 2005, 117 (28): 4429–4432. doi:10.1002/ange.200500703.
- ^ DNA walkers: Bath, Jonathan; Green, Simon J.; Turberfield, Andrew J. A free-running DNA motor powered by a nicking enzyme. Angewandte Chemie International Edition. 11 July 2005, 44 (28): 4358–4361. doi:10.1002/anie.200501262.
- ^ Functional DNA walkers: Lund, Kyle; Manzo, Anthony J.; Dabby, Nadine; Michelotti, Nicole; Johnson-Buck, Alexander; Nangreave, Jeanette; Taylor, Steven; Pei, Renjun; Stojanovic, Milan N.; Walter Nils G.; Winfree, Erik; Yan, Hao. Molecular robots guided by prescriptive landscapes. Nature. 13 May 2010, 465 (7295): 206–210. Bibcode:2010Natur.465..206L. PMC 2907518
 . PMID 20463735. doi:10.1038/nature09012. (英文)
. PMID 20463735. doi:10.1038/nature09012. (英文)
- ^ Functional DNA walkers: He, Yu; Liu, David R. Autonomous multistep organic synthesis in a single isothermal solution mediated by a DNA walker. Nature Nanotechnology. November 2010, 5 (11): 778–782. Bibcode:2010NatNa...5..778H. PMC 2974042
 . PMID 20935654. doi:10.1038/nnano.2010.190.(英文)
. PMID 20935654. doi:10.1038/nnano.2010.190.(英文)
- ^ Strand displacement cascades: Georg Seelig, David Soloveichik, David Yu Zhang, Erik Winfree. Enzyme-Free Nucleic Acid Logic Circuits. Science. 2006-12-08, 314 (5805): 1585–1588 [2018-04-02]. ISSN 0036-8075. doi:10.1126/science.1132493. (原始內容存檔於2018-03-06) (英語).
- ^ Strand displacement cascades: Lulu Qian, Erik Winfree. Scaling Up Digital Circuit Computation with DNA Strand Displacement Cascades. Science. 2011-06-03, 332 (6034): 1196–1201 [2018-04-02]. ISSN 0036-8075. doi:10.1126/science.1200520. (原始內容存檔於2018-02-14) (英語).
- ^ Applications: Rietman, Edward A. Molecular engineering of nanosystems. Springer. 2001: 209–212 [17 April 2011]. ISBN 978-0-387-98988-4.(英文)
- ^ Applications: Jungmann, Ralf; Renner, Stephan; Simmel, Friedrich C. From DNA nanotechnology to synthetic biology. HFSP journal. March 2008, 2 (2): 99–109. PMC 2645571
 . PMID 19404476. doi:10.2976/1.2896331.(英文)
. PMID 19404476. doi:10.2976/1.2896331.(英文)
- ^ Methods: Andrew Ellington, Jack D. Pollard. Synthesis and Purification of Oligonucleotides 42. Wiley-Blackwell. 1998-04-01 [2018-04-02]. doi:10.1002/0471142727.mb0211s42 (英語).
- ^ Methods: Andrew Ellington, Jack D. Pollard. Purification of Oligonucleotides Using Denaturing Polyacrylamide Gel Electrophoresis 42. Wiley-Blackwell. 1998-04-01 [2018-04-02]. doi:10.1002/0471142727.mb0212s42 (英語).
- ^ Methods: Sean R. Gallagher, Philippe Desjardins. Quantitation of Nucleic Acids and Proteins. doi:10.1002/9780470089941.et0202s5.(英文)
- ^ Methods: Joanne Chory, Jack D. Pollard. Separation of Small DNA Fragments by Conventional Gel Electrophoresis 47. Wiley-Blackwell. 1999-07-01 [2018-04-02]. doi:10.1002/0471142727.mb0207s47 (英語).
- ^ Methods: Nils G. Walter. Probing RNA Structural Dynamics and Function by Fluorescence Resonance Energy Transfer (FRET) 11. Wiley-Blackwell. 2002-12-01: 11.10.1–11.10.23 [2018-04-02]. doi:10.1002/0471142700.nc1110s11 (英語).
- ^ Methods: Chenxiang Lin, Yonggang Ke, Rahul Chhabra, Jaswinder Sharma, Yan Liu, Hao Yan. Synthesis and Characterization of Self-Assembled DNA Nanostructures. Humana Press. 2011: 1–11 [2018-04-02]. ISBN 9781617791413. doi:10.1007/978-1-61779-142-0_1. (原始內容存檔於2018-06-13) (英語).
- ^ Methods: Bloomfield, Victor A.; Crothers, Donald M., Tinoco, Jr., Ignacio. Nucleic acids: structures, properties, and functions. Sausalito, Calif: University Science Books. 2000: 84–86, 396–407. ISBN 0-935702-49-0.
擴展閱讀
[編輯]General:
- Seeman, Nadrian C. Nanotechnology and the double helix. Scientific American. June 2004, 290 (6): 64–75 [2012-08-28]. PMID 15195395. doi:10.1038/scientificamerican0604-64. (原始內容存檔於2013-11-10).—An article written for laypeople by the founder of the field
- Seeman, Nadrian C. Structural DNA nanotechnology: growing along with Nano Letters. Nano Letters. 9 June 2010, 10 (6): 1971–1978. Bibcode:2010NanoL..10.1971S. PMC 2901229
 . PMID 20486672. doi:10.1021/nl101262u.—A review of results in the period 2001–2010
. PMID 20486672. doi:10.1021/nl101262u.—A review of results in the period 2001–2010 - Seeman, Nadrian C. Nanomaterials based on DNA. Annual Review of Biochemistry. 2010, 79: 65–87. PMID 20222824. doi:10.1146/annurev-biochem-060308-102244.—A more comprehensive review including both old and new results in the field
- Service, Robert F. DNA nanotechnology grows up. Science. 3 June 2011, 332 (6034): 1140–1143. doi:10.1126/science.332.6034.1140. and doi:10.1126/science.332.6034.1142.—A news article focusing on the history of the field and development of new applications
- Zadegan, Reza M.; Norton, Michael L. Structural DNA Nanotechnology: From Design to Applications. Int. J. Mol. Sci. June 2012, 13 (6): 7149–7162 [2012-08-28]. PMC 3397516
 . PMID 22837684. doi:10.3390/ijms13067149. (原始內容存檔於2012-11-14).—A very recent and comprehensive review in the field
. PMID 22837684. doi:10.3390/ijms13067149. (原始內容存檔於2012-11-14).—A very recent and comprehensive review in the field
Specific subfields:
- Bath, Jonathan; Turberfield, Andrew J. DNA nanomachines. Nature Nanotechnology. 5 May 2007, 2 (5): 275–284. Bibcode:2007NatNa...2..275B. PMID 18654284. doi:10.1038/nnano.2007.104.—A review of nucleic acid nanomechanical devices
- Feldkamp, Udo; Niemeyer, Christof M. Rational design of DNA nanoarchitectures. Angewandte Chemie International Edition. 13 March 2006, 45 (12): 1856–76. PMID 16470892. doi:10.1002/anie.200502358.—A review coming from the viewpoint of secondary structure design
- Lin, Chenxiang; Liu, Yan; Rinker, Sherri; Yan, Hao. DNA tile based self-assembly: building complex nanoarchitectures. ChemPhysChem. 11 August 2006, 7 (8): 1641–1647. PMID 16832805. doi:10.1002/cphc.200600260.—A minireview specifically focusing on tile-based assembly
- Zhang, David Yu; Seelig, Georg. Dynamic DNA nanotechnology using strand-displacement reactions. Nature Chemistry. February 2011, 3 (2): 103–113. Bibcode:2011NatCh...3..103Z. PMID 21258382. doi:10.1038/nchem.957.—A review of DNA systems making use of strand displacement mechanisms
外部連結
[編輯]- International Society for Nanoscale Science, Computation and Engineering(頁面存檔備份,存於網際網路檔案館)
- What is Bionanotechnology?(頁面存檔備份,存於網際網路檔案館)—a video introduction to DNA nanotechnology




