核酸三级结构[编辑]
核酸三级结构是核酸聚合物的三维形状[1]。 RNA和DNA分子具有从分子识别到催化的各种功能。 这些功能需要精确的三维三级结构。 虽然这种结构多样且看似复杂,但它们由重复的,易于识别的三级结构基序组成,用作分子构建模块。 RNA和DNA三级结构的一些最常见的基序如下所述,但该信息基于有限数量的解析结构。
螺旋结构
[编辑]
双螺旋
[编辑]双螺旋是生物DNA的主要三级结构,也是RNA的可能结构。据信在自然界中发现了三种DNA构象,即A-DNA,B-DNA,和Z-DNA。 詹姆斯·杜威·沃森(James D. Watson)和弗朗西斯·克里克(Francis Crick)描述的“B”形式被认为在细胞中占主导地位[2]。詹姆斯·D·沃森和弗朗西斯·克里克将这种结构描述为半径为10Å且间距为34Å的双螺旋,每隔10 bp序列绕其轴旋转一圈[3]。双螺旋在溶液中每10.4-10.5碱基对围绕其轴旋转一圈。这种扭曲频率(称为螺旋节距)很大程度上取决于每个基座对链中的邻居施加的堆叠力。双螺旋RNA采用类似于A型结构的构象。
其他构造是可能的;事实上,现在只有字母F,Q,U,V和Y可用于描述将来可能出现的任何新的DNA结构[4][5]。然而,大多数这些形式是合成产生的,并且在天然存在的生物系统中未观察到。
主要的和次要的凹槽三联体
[编辑]次要的凹槽三股螺旋是普遍存在的RNA结构基序。 因为与次要的凹槽的相互作用通常由核糖的2'-OH介导,所以这种RNA基序看起来与其DNA等同物非常不同。 次要的凹槽三股螺旋的最常见的例子是A-次要基序,或腺苷碱基插入次要的凹槽(见上文)。 然而,该基序不限于腺苷,因为还观察到其他核碱基与RNA次要的凹槽相互作用。
次要的凹槽为插入的底座提供了近乎完美的补充。 这允许最佳的范德华接触,广泛的氢键的和疏水的表面埋藏,并产生高能量有利的相互作用。 因为次要的凹槽三元组能够稳定地包装自由环和螺旋,它们是大核糖核苷酸结构中的关键元件,包括I组内含子[10], II组内含子[11],和核糖体。
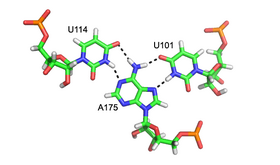
尽管标准A型RNA的主要的凹槽相当窄,因此与次要的凹槽相比,三重相互作用的可用性较小,但在几种RNA结构中可观察到主要的凹槽三重相互作用。 这些结构由碱基对和Hoogsteen相互作用的几种组合组成。 例如,在50S核糖体中观察到的GGC三联体(GGC氨基(N-2)-N-7,亚氨基 - 羰基,羰基 - 氨基(N-4); 沃森-克里克),由沃森-克里克型GC组成 配对和进入的G,其形成在规范配对中涉及的两个碱基之间的氢键相互作用的伪Hoogsteen网络[8]。其他值得注意的主要的凹槽三联体的例子包括(i)左图中所示的II族内含子的催化核心[6],(ii)在人类的端粒酶RNA組分中观察到的催化必需的三股螺旋[7],和(iii)SAM -II riboswitch[12]。
来自Hoogsteen的三股螺旋DNA也可能在B型DNA的主要沟槽中逆转Hoogsteen氢键。
四联体
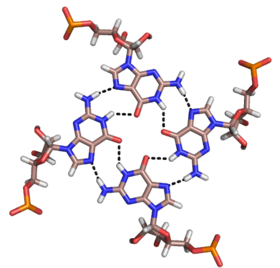
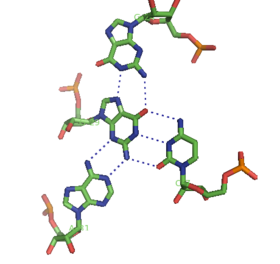
[编辑]除了双股螺旋和上述三股螺旋之外,RNA和DNA也可以形成四股螺旋的四联体。 RNA碱基四联体有多种结构。 通过Hoogsteen氢键,四个连续的鸟嘌呤残基可在RNA中形成四股螺旋,形成“Hoogsteen环”(见图)。 G-C和A-U对也可以形成碱基四联体,其中沃森-克里克(Watson-Crick)配对和次要的凹槽中的非规范配对的组合[13]。
孔雀石绿色的适体的核心也是一种具有不同氢键模式的碱基四联体(见图).[9]。 四联体可以连续重复几次,产生非常稳定的结构。
参阅
[编辑]参考资料
[编辑]- ^ 國際純化學和應用化學聯合會,化學術語概略,第二版。(金皮書)(1997)。在線校正版: (2006–) "tertiary structure"。doi:10.1351/goldbook.T06282
- ^ Richmond TJ, Davey CA. The structure of DNA in the nucleosome core. Nature. May 2003, 423 (6936): 145–50. Bibcode:2003Natur.423..145R. PMID 12736678. doi:10.1038/nature01595.
- ^ Watson JD & Crick FH. Molecular structure of nucleic acids; a structure for deoxyribose nucleic acid (PDF). Nature. April 1953, 171 (4356): 737–8 [2018-11-09]. Bibcode:1953Natur.171..737W. PMID 13054692. doi:10.1038/171737a0. (原始内容存档 (PDF)于2007-02-04).
- ^ Bansal M. DNA structure: Revisiting the Watson-Crick double helix. Current Science. 2003, 85 (11): 1556–1563.
- ^ Ghosh A, Bansal M. A glossary of DNA structures from A to Z. Acta Crystallogr D. 2003, 59 (4): 620–626. PMID 12657780. doi:10.1107/S0907444903003251.
- ^ 引用错误:没有为名为
Toor08的参考文献提供内容 - ^ 跳转到: 7.0 7.1 PDB 2K95; Kim NK, Zhang Q, Zhou J, Theimer CA, Peterson RD, Feigon J. Solution structure and dynamics of the wild-type pseudoknot of human telomerase RNA. J. Mol. Biol. December 2008, 384 (5): 1249–61. PMC 2660571
 . PMID 18950640. doi:10.1016/j.jmb.2008.10.005.; rendered with PyMOL (页面存档备份,存于互联网档案馆)
. PMID 18950640. doi:10.1016/j.jmb.2008.10.005.; rendered with PyMOL (页面存档备份,存于互联网档案馆)
- ^ 跳转到: 8.0 8.1 PDB 1RAU; Cheong C, Moore PB. Solution structure of an unusually stable RNA tetraplex containing G- and U-quartet structures. Biochemistry. September 1992, 31 (36): 8406–14. PMID 1382577. doi:10.1021/bi00151a003.; rendered with PyMOL (页面存档备份,存于互联网档案馆)
- ^ 跳转到: 9.0 9.1 PDB 1FIT; Baugh C, Grate D, Wilson C. 2.8 A crystal structure of the malachite green aptamer. J. Mol. Biol. August 2000, 301 (1): 117–28. PMID 10926496. doi:10.1006/jmbi.2000.3951.; rendered with PyMOL (页面存档备份,存于互联网档案馆)
- ^ Szewczak AA, Ortoleva-Donnelly L, Ryder SP, Moncoeur E, Strobel SA. A minor groove RNA triple helix within the catalytic core of a group I intron. Nat. Struct. Biol. December 1998, 5 (12): 1037–42. PMID 9846872. doi:10.1038/4146.
- ^ Boudvillain M, de Lencastre A, Pyle AM. A tertiary interaction that links active-site domains to the 5' splice site of a group II intron. Nature. July 2000, 406 (6793): 315–8. PMID 10917534. doi:10.1038/35018589.
- ^ Gilbert SD, Rambo RP, Van Tyne D, Batey RT. Structure of the SAM-II riboswitch bound to S-adenosylmethionine. Nat. Struct. Mol. Biol. February 2008, 15 (2): 177–82. PMID 18204466. doi:10.1038/nsmb.1371.
- ^ Batey RT, Gilbert SD, Montange RK. Structure of a natural guanine-responsive riboswitch complexed with the metabolite hypoxanthine. Nature. November 2004, 432 (7015): 411–5. Bibcode:2004Natur.432..411B. PMID 15549109. doi:10.1038/nature03037.