DNA纳米技术:修订间差异
小 →纳米机械装置 |
→应用: References |
||
| 第107行: | 第107行: | ||
==应用== |
==应用== |
||
DNA纳米科技提供了设计构建复杂结构并精确控制纳米特性的一种方法。这个领域开始应用在结构生物学与生物物理学中解决基础科学问题。对这类应用最早的展望是在[[结晶学]],孤立的分子很难结晶化,把分子布置三维核酸[[晶格]]内,以确定它们的结构。另一种应用是在蛋白质[[核磁共振波谱学]]中的残留偶极耦合实验中用DNA折纸棒来代替[[液晶]]。用DNA折纸术是非常有好处的,因为不像液晶,DNA折纸棒可以承受使膜蛋白悬浮的洗涤剂。DNA 行走者(DNA walkers)被用作纳米组合线来移动纳米粒子,指导化学合成。不仅如此,DNA折纸结构也促进了[[酶]]功能和[[蛋白质]]折叠方面的研究。 |
DNA纳米科技提供了设计构建复杂结构并精确控制纳米特性的一种方法。这个领域开始应用在结构生物学与生物物理学中解决基础科学问题。对这类应用最早的展望是在[[结晶学]],孤立的分子很难结晶化,把分子布置三维核酸[[晶格]]内,以确定它们的结构。另一种应用是在蛋白质[[核磁共振波谱学]]中的残留偶极耦合实验中用DNA折纸棒来代替[[液晶]]。用DNA折纸术是非常有好处的,因为不像液晶,DNA折纸棒可以承受使膜蛋白悬浮的洗涤剂。DNA 行走者(DNA walkers)被用作纳米组合线来移动纳米粒子,指导化学合成。不仅如此,DNA折纸结构也促进了[[酶]]功能和[[蛋白质]]折叠方面的研究。<ref name="SDNreview" /><ref name="growsup" /> |
||
DNA纳米科技正朝潜在的实际应用迈步。核酸组列安置其他分子的能力暗示了其在分子电子学上的应用前景。核酸结构可以被用作分子电子单位(如分子导线(molecular wires))组合的模板,提供对安置进行控制的方法,类似于一个电路实验板。DNA纳米技术被比作可编程物质(programmable matter),因为其材料的耦合计算。 |
DNA纳米科技正朝潜在的实际应用迈步。核酸组列安置其他分子的能力暗示了其在分子电子学上的应用前景。核酸结构可以被用作分子电子单位(如分子导线(molecular wires))组合的模板,提供对安置进行控制的方法,类似于一个电路实验板。<ref name="SDNreview" /><ref name="ShihYanChallenges" />DNA纳米技术被比作可编程物质(programmable matter),因为其材料的耦合计算。<ref>'''Applications:''' {{cite book|last=Rietman|first=Edward A.|title=Molecular engineering of nanosystems|year=2001|publisher=Springer|isbn=978-0-387-98988-4|pages=209–212|url=http://books.google.com/books?id=ga2DKYCm7xMC&pg=PA209|accessdate=17 April 2011}}</ref> |
||
DNA纳米科技在纳米机械方面也有潜在的应用。比如利用其以生物相容性的计算制药,使之能够靶向给药(targeted drug delivery)。一个现在正被研究的系统用了一个中空的DNA盒子,其中包着可以引发细胞凋亡或死亡的蛋白质,而只有靠近癌细胞时,盒子才会打开,释放出蛋白质。人们对在[[细胞]]中表达人工结构怀着极大兴趣,最有可能使用转录RNA进行组装,尽管仍不知道这些合成结构是否能有效地折叠或聚集在细胞质中。 |
DNA纳米科技在纳米机械方面也有潜在的应用。比如利用其以生物相容性的计算制药,使之能够靶向给药(targeted drug delivery)。一个现在正被研究的系统用了一个中空的DNA盒子,其中包着可以引发细胞凋亡或死亡的蛋白质,而只有靠近癌细胞时,盒子才会打开,释放出蛋白质。<ref name="growsup" /><ref>'''Applications:''' {{cite journal |pmid=19404476 |month=March |year=2008 |last1=Jungmann |first1=Ralf |last2=Renner |first2=Stephan |last3=Simmel |first3=Friedrich C. |title=From DNA nanotechnology to synthetic biology |volume=2 |issue=2 |pages=99–109 |doi=10.2976/1.2896331 |pmc=2645571 |journal=HFSP journal}}</ref>人们对在[[细胞]]中表达人工结构怀着极大兴趣,最有可能使用转录RNA进行组装,尽管仍不知道这些合成结构是否能有效地折叠或聚集在细胞质中。<ref name="ShihYanChallenges" /> |
||
==材料与方法== |
==材料与方法== |
||
2012年8月28日 (二) 06:01的版本
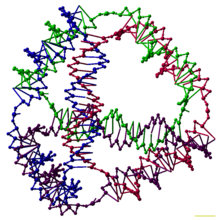
DNA奈米科技(DNA nanotechnology)專門研究利用DNA或其他核酸的分子性質(如自組裝的特性),來建構出可操控的新型奈米尺度結構或機械。在这个领域,核酸被用作非生物的材料而不是在活细胞中那样作为遗传信息的载体。严格的核酸碱基配对法则(使链上特定的碱基列相互连接以形成牢固的双螺旋结构)使这一技术成为可能。这一技术允许合理的碱基链设计,从而严格地组合形成具有精密控制的纳米级特性的复杂的目标结构。DNA是常使用的优势材料,但包括其他核酸如RNA和PNA(肽核酸)也被用来构造结构,所以偶尔也用“核酸纳米科技”来概括这个领域。[2][3]
DNA纳米科技概念的基础最先由纳德里安•西曼(Nadrian Seeman)在八十年代早期阐述,在2000年后开始引起广泛的关注。这一领域的研究者已经构建了静止结构如二维和三维晶体结构、毫微管、多面体和其他任意的造型;和功能结构如纳米机器和DNA计算机。一些组建方法被用来构建拼装结构、折叠结构和动态可重构结构。现在,这种科技开始被用作解决在结构生物学和生物物理学中基础科学问题的工具;同时也被应用在结晶学和光谱学中来测定蛋白质结构。这项技术在分子电子学(molecular scale electronics)和纳米医学(nanomedicine)中的应用仍在研究中。
基础概念


核酸的特性
纳米科技常被限定为在小于100纳米的尺度下对材料和物体的研究。DNA 纳米科技是自下而上分子自组装的一个例子,即分子成分自发地组成稳定结构;这些结构的特殊形态是由设计者所挑选的成分的物化特性所引发的。[6]在DNA纳米科技中,构建成分是核酸链,如DNA。DNA很适合纳米级的构造,因为一条DNA双螺旋直径为2nm,螺旋重复长度为3.5nm。核酸比其他材料更适合构造结构的主要特性是两条核酸链间简单的碱基配对法则,通过碱基连接可以形成一个具体明确的结构。这使核酸结构的组合容易通过核酸设计得到控制。这种特性在其他被用于纳米技术的材料中并不存在(包括蛋白质和纳米颗粒)。[7]
核酸分子的结构由含不同碱基的核苷酸的排列顺序决定。在DNA中,四个碱基为腺嘌呤、胞嘧啶、鸟嘌呤和胸腺嘧啶。如果核酸的两条链是互补的,它们将相互连接形成双螺旋结构(A-T,C-G)。[7][8]因为正确的碱基配对是积极有利的,在大多数情况下,核酸链相互连接时,都要使正确配对的碱基数目最大化。链中碱基的排列决定了连接的方式和整体结构,这简单可控。在DNA纳米技术中,研究者设计合理的碱基序列,从而使得整条链按照研究者希望的那样组合起来。[5][7]
分支
DNA纳米科技有时被分为两个有所重叠的领域:结构DNA纳米技术(structural DNA nanotechnology)和动态DNA纳米技术(dynamic DNA nanotechnology)。结构DNA纳米技术(有时缩略为SDN)主要方向是合成核酸材料等合成物并组合成稳定平衡的最终形态。[9][10]
而动态DNA纳米技术的主要方向是研究具有不平衡性状(如在物化条件的诱导下具有重组能力)的合成物。一些合成物如核酸纳米机械(nucleic acid nanomechanical devices)同时具有这两个分支的特性。在结构DNA纳米技术中构建的合成物会有拓扑分支的核酸结构。(大多数生物DNA是不含分支的双螺旋结构。)一个最简单的分支结构是在一个节点上产生四条分支,这包含四股独立的DNA链。不像自然的霍雷迪交叉(Holiday junctions),在这个人工节点上每条分支都有不同的碱基序列,因此这个节点被固定在某个位置。多个节点可以在同一个合成物中被组合起来,如广泛使用的双交叉砖(double-crossover (DX)motif),它由两条平行的双螺旋链和独立的在其间交叉的链组成。其交叉于两个点,每个点都是一个四支节点,但是与一个可变的四支节点不同,它被约束到单一的方向,使DX砖作为对更大的DNA合成物的结构障碍。[7][5]
动态DNA纳米技术运用了一种叫做toehold-mediated strand displacement的机制使核酸合成物对新核酸链的添加做出反应进行重组。在这个反应中,移入的链与双链合成物中一条链上的点位进行连接,并在分支迁移过程中置换掉初始合成物中的一条链。也就是合成物中的一条链被另外的链所替换。[9]重组结构与机械也可用功能核酸制造,如脱氧核酶、核酶或核酸适体。[11]
设计
DNA的纳米结构必须被合理设计,单条核酸链才会被组合成目标结构。这个过程通常需要对目标结构或功能的详述。然后确定目标合成物的二级结构即对结构内核酸链的安置和连接点位。最后是一级结构的设计即对每条核酸链碱基顺序的安排。[12][13]
结构设计
设计核酸纳米结构的第一步是决定如何用核酸链构建目标结构。这一步决定了其二级结构和连接点位。[12]有如下一些方法:
- 拼接结构:这种方法打破部分化学键将目标结构分解成小单元。这常被用来做点阵,但也被用来实施系统的自组,使之成为DNA计算的平台。这是从90年代中期DNA折纸法发展以来十多年的主导设计策略。[12][14]
- 折叠结构:折叠法是用一条长链构造纳米结构。这条长链可以有设计好的碱基序列而自身折叠或者用短一些的链“订”在一起,折叠成目标结构。这后一种方法叫做DNA折纸(DNA origami)。通过这种方法,构建纳米级的二维或三维形态也得以实现(见下面的非连续结构)[15][16]
- 动态组合:这种方法直接控制DNA自组的动力,除了中产物外要分析反应中所有的中间步骤。用采用了发夹结构的原始材料;这些发生级联反应以一个特定的顺序形成最终构造。这种方法有在恒定温度下进行的优势。这不同于热力学方法,它不需要热退火步骤来使之形成目标结构。[15][17]
序列设计
在用上述方法设计了目标合成物的二级结构后,核苷酸的序列必须要确定出。许多方法都以目标结构具有最低能量为目的设计序列,因为这在热力学上是最有利的,而拆卸的结构具有更高的能量,因此不有利。这样做是可以通过更快的、简单的、启发性的方法如序列对称最小化,或使用一个完整的最邻近(nearest-neighbor)热力学模型,这更精确但更慢并且需要更密集的计算。几何模型用来测定纳米结构的三级结构以保证合成物不会过度紧密。[13][18]
核酸设计与蛋白质设计有相似的目标。在两者中,单体的序列被设计成有利于目标结构而不利于其他结构。核酸设计比蛋白质设计的计算要更简单,因为简单的碱基配对法则足够预测结构的能量支持(energetic favorability),并且关于总体上结构的三维折叠的细节信息是不需要的。这允许使用简单的启发式方法产生稳固的实验性设计。然而,核酸结构在功能上多样性比蛋白质弱,因为有更强的折叠成合成结构,并且与20种氨基酸相比,四种碱基缺少化学多样性。[18]
结构DNA纳米技术

结构DNA纳米技术(有时缩写为SDN),着重于对核酸合成物的分析与描述。核酸的双螺旋是一个稳固的三维几何结构,这使得预测和设计更为复杂的核酸合成物成为可能。许多这样的结构已经被创造出来包括二维、三维结构,周期性的、非周期性的和离散结构。[10]
扩展晶格
小的核酸合成物可以配上粘性末端,合成更大的二维周期性晶格(含由个体砖组建的特殊的密铺型)。[10]最早的一个例子是用DX砖作为基础砖,每个砖都包含四个设计好的粘性末端,以使DX砖可以合成二维平面结构。这基本上是刚性的DNA二维晶格。[22][23]二维组列也可以用其他形制造如霍雷迪交叉菱形晶格和各种以DX砖为基础的组列。[24][25]上面两幅图便是周期性晶格的例子。
二维组列可以表现出非周期性结构,它们的组合能够执行运算,展示出DNA计算的一种形式。[26]DX砖可以通过选择带粘性末端的序列充当王氏砖,并执行运算DX组列。能够编码逻辑异或程序的DX组列已被验证;这使DNA组列可以实施一个细胞自动机,生成一个不规则碎片形(塞平斯基垫片结构)。右边第三个图片展现了这个组列的形式。[21]另一个系统有二进制计数功能,它的增长意味着二进制数的增加。[27]
DX组列已经能被做出直径4-20nm的中空纳米管,这主要是二维晶格自身卷起。[12]这些DNA纳米管在形状大小上和碳纳米管有一点相似,但缺少碳纳米管的电导。DNA纳米管更容易被修改、被连接到其他结构。构建DNA纳米碳管的其中一个方案是用一个DX砖晶格,它自身卷起成管。[28]另一种方法是用单链砖使圆周被指定在一个简单的模块化的形式,这个管的刚性是一个新生特性。[29]
用DNA创建三维晶格是DNA纳米科技最早的目标,但这很难实现。最终在2009年,报道称成功地使用建立在无尺寸限制结构的概念上的图形创建出三维结构并在紧张压缩间达到平衡。[26][30]
离散结构
研究者已经分析了一些与多面体相关的三维DNA合成物,每个都与多面体有联通性,比如立方体或八面体,这意味着DNA双链是多面体的棱而每个顶点都是一个DNA节点。[15]早期对DNA多面体的验证工作量很大,需要复杂的结扎和固相结合的步骤来建造连续的多面体。[31]随后的工作使多面体的分析变得简单。[32]这些包括一个用一条长链制造DNA八面体,和一步用四条DNA链制成一个四面体。(见上图)[1]
任意的纳米结构、不规则的形状通常用DNA折纸法制成。这些结构有一条病毒链作为支架,这被用计算设计的短链订成目标形状。这种方法有容易设计的优势,因为碱基序列是被支架碱基序列所预先确定的,并且不需要高的链纯度和精确的化学计量。DNA折纸法首先在二维形中使用,比如表情符号和粗略的北美地图。[15][16]刚性的三维结构可以通过使用安排成蜂窝状的DNA双螺旋制造;[33]二维平面可以被折叠成中空的三维形状,类似于一个箱子。这些可以被编码为对于刺激做出反应,使它们作为分子笼。[34][35]
模板组装
核酸分子可以吸收其他分子如蛋白质金属纳米离子、量子点(quantum dots)、富勒烯。这使得构建具有多种功能的机械变得易行。用核酸结构的自组来作为附加粒子自组的样板,控制它们的位置,并在一些情况下定位。[15][36]许多方案使用共价结合的办法,用酰胺或硫醇为官能团的寡核酸链绑定异质粒子。[37]这种共价结合的办法已经被用来把金纳米粒子和链霉亲和素分子固定在DX砖组列上。[38]有一种非共价结合法用Dervan聚酰胺。[39]碳纳米管以某种形状被连接到DNA组列上充当分子电子机器——碳纳米管场效应晶体管。[40]另外,还有核酸金属化法即用金属代替核酸呈现最初的核酸结构。——用核酸纳米结构作为模具,将形状转化为固体型。[41][42]
动态DNA纳米技术

动态DNA纳米技术着重于建造具有动态功能(如计算和机械运动)的与整体结构相关的核酸系统。结构DNA纳米技术和动态DNA纳米技术有所重叠。因为结构可以通过退火和动态重构形成或者首先动态组合。[15][43]
纳米机械装置
人们已经制造出随着一些外界刺激改变造型的DNA合成物——这是纳米机器人的一种形式。这些结构最初都是以与构建静止结构相同的方式形成的;但经过设计,于是在最初的形成后能够动态重构。[9][43]最早的这样的机械利用了B-DNA与Z-DNA间的转变来对在缓冲环境下通过扭转运动做出反应。[44]然而,这倚赖于缓冲液的条件使所有的机械在同一时间改变状态。后续的系统可以根据控制链的存在改变状态,使多种机械可在溶液中独立地运转。这样系统的一个例子是“分子镊”设计。[45]它有开和闭的形式,在旋转运动过程中从平行交叉构造(paranemic-crossover (PX))转为双节构造(double-junction (JX2))。[46]另有一种二维组列可以动态地响应控制连进行扩张或收缩。[47]人们已造出可以动态开关,能作为“分子笼”在打开时释放功能“货物”。[34][48][49]
DNA行走者(DNA walkers)是一类沿直线轨道运动的核酸纳米机器。大量的方案已被验证。[43]其中一个方案是用需人工添加序列的控制链控制行走者沿轨迹的移动。[50][51]另一个方案是利用限制酶或脱氧核酶解开链,使行走者前移,这种方法的优点是自动运行。[52][53]后来一种系统可以在二维平面而不是一条直线上运动,并且被证实有选择性地获取和移动分子货物的功能。[54]另外,一个直线行走者被证实在其移动的过程中会进行DNA合成。[55]
链置换级联反应
不论计算或结构目的链置换级联反应都可以使用。单独的链链置换反应包括对一些引发链的存在做出反应产生新序列。许多这样的反应被连到级联上,一个反应新产生的序列可以引起另一个链置换级联反应。这反过来又促进了化学反应网络的建设,表现出复杂的计算和信息处理能力。通过新键的形成和从分解反应中获得的熵,级联变得积极有利。链置换级联反应允许组合与计算过程中的等温操作(这与传统核酸组合要求热退火工序相反)。它们也支持催化功能,小于一当量的引发剂就可以使反应完成。[9][17]
链置换合成物可以用来制造能执行复杂运算的分子逻辑门。不像用电流作为输入和输出的传统的电子计算机,分子计算机用特定化学物质的浓度作为信号。在核酸链置换电路中,信号是核酸与其他链的连接与断开释放或吸收的核酸。这种方法已被用来做与、或、非门。[56]最近,一个四位电路被验证可以做0-15整数平方根的计算(用包含130条DNA的逻辑门)。[57]
链置换级联反应的另一个作用是制造动态组合结构。这些用发夹结构作为反应物,当输入的链连接时,新序列会在同一分子上而不是分解。这使新打开的发夹添加到合成物中。这种方法已被用来做简单的结构如三或四支节点和树状物。[17]
应用
DNA纳米科技提供了设计构建复杂结构并精确控制纳米特性的一种方法。这个领域开始应用在结构生物学与生物物理学中解决基础科学问题。对这类应用最早的展望是在结晶学,孤立的分子很难结晶化,把分子布置三维核酸晶格内,以确定它们的结构。另一种应用是在蛋白质核磁共振波谱学中的残留偶极耦合实验中用DNA折纸棒来代替液晶。用DNA折纸术是非常有好处的,因为不像液晶,DNA折纸棒可以承受使膜蛋白悬浮的洗涤剂。DNA 行走者(DNA walkers)被用作纳米组合线来移动纳米粒子,指导化学合成。不仅如此,DNA折纸结构也促进了酶功能和蛋白质折叠方面的研究。[10][58]
DNA纳米科技正朝潜在的实际应用迈步。核酸组列安置其他分子的能力暗示了其在分子电子学上的应用前景。核酸结构可以被用作分子电子单位(如分子导线(molecular wires))组合的模板,提供对安置进行控制的方法,类似于一个电路实验板。[10][15]DNA纳米技术被比作可编程物质(programmable matter),因为其材料的耦合计算。[59]
DNA纳米科技在纳米机械方面也有潜在的应用。比如利用其以生物相容性的计算制药,使之能够靶向给药(targeted drug delivery)。一个现在正被研究的系统用了一个中空的DNA盒子,其中包着可以引发细胞凋亡或死亡的蛋白质,而只有靠近癌细胞时,盒子才会打开,释放出蛋白质。[58][60]人们对在细胞中表达人工结构怀着极大兴趣,最有可能使用转录RNA进行组装,尽管仍不知道这些合成结构是否能有效地折叠或聚集在细胞质中。[15]
材料与方法

DNA链是用分子建模和热力学建模软件计算设计的。核酸用标准的寡核苷酸合成法合成,通常用寡核苷酸合成器自动进行合成,自定义序列的链是市售的。如果需要的话,链可以通过变性凝胶电泳纯化,精确的浓度可用紫外线吸收光谱来定量测定。
完全形成的目标结构可用非变性凝胶电泳检验,将会得到关于核酸合成物形态大小的信息。电泳迁移率变动分析可以评估一个结构是否包含所有的所需链。荧光标记和荧光共振能量转移(FRET)有时用一表示合成物结构的特性。
核酸结构可以直接用原子力显微镜看到,这很适合延伸的二维结构,但对三维结构没有多大用处,因为显微镜尖端会与脆弱的核酸结构相互作用;因此透射电子显微镜和低温电子显微镜常用以观察三维结构。延伸的三维晶格用X光晶体学分析。
历史
DNA 纳米科技概念的基础最先由纳德里安•西曼(Nadrian Seeman) 在八十年代早期阐述。西曼的最初目的是创建一个三维的DNA晶格为其他大分子确定方向,这将去除获得纯净晶体的复杂过程,简化晶体学研究。据说他在意识到莫里茨•科内利斯•埃舍尔(M. C. Escher)的木刻Depth与大批的六节点DNA间的共同点后,产生了这个想法。在当时,一些自然地有分支的DNA结构已被人们知晓包括复制叉和可移动的霍雷迪交叉(Holiday junctions),但西曼认为不可动的核酸节点可以通过正确的碱基序列设计消除组合分子的对称性来制造。那样这些不可动节点在原则上可以组合成稳固的晶格。第一篇阐明这个体系的论文于1982年发表,第一份试验证明于次年刊出。
在1991年,西曼的实验室发布了一份对用DNA制成的立方体(第一个人工合成的三维核酸纳米结构)的分析报告。他因此获得1995年费曼纳米技术奖(Feynman Prize in Nanotechnology)。DNA截面八角体也随之诞生。然而,人们很快发现这些以可变节点作为顶点的多边形结构对构造伸展的三维晶格来说不够牢固。西曼发展了更为牢固的双交叉形(DX),在1998年与Erik Winfree的合作中发布了用DX拼接成的二维晶格。这些拼接结构具有实施DNA计算(DNA computing)的能力。这一点已由Winfree和Paul Rothemund在他们2004年关于塞平斯基垫片结构(Sierpinski gasket)的论文中证明,他们因此分享了2006年费曼纳米技术奖。Winfree的主要观点是DX砖可以当做王氏砖(Wang tiles)使用,也就是说他们的组合可进行计算。对三维晶格的分析最终由西曼在2009年发表,为完成它西曼花了大约30年。
在2000年代设计的DNA结构的新功能陆续被发现。第一个DNA纳米机器—会对输入做出反应而改变其形态-在1999年由西曼验证。次年,Bernard Yurke验证了一个有提升的核酸机器。更进一步的,这被翻译成机械运动,并在2004和2005年一些DNA行走器件被西曼、Niles Pierce、 Andrew Turberfield和Chengde Mao所验证。用DNA组列构建其他分子的模板(如纳米粒子、蛋白质等)的构想最早由Bruche Robinson 和西曼在1987年提出,在2006、2007年被Hao Yan、Peter Dervan和Thomas Labean验证。
在2006年,Rothemund第一次验证了DNA折纸技术可以简单地构建出稳固的具有任意造型的结构。Rothemund设想这种用多条短链的方法是介于西曼晶格(用多条短链)和William Shih的DNA八面体(主要用一条长链)的概念性中产物。Rothemund的DNA折纸法是用一条长链和许多短链配合折叠。这种方法使构建更大的结构变得可行并降低了对设计和分析的技术要求。DNA折纸术在2006年3月15日登上了《自然》杂志的封面。随Rothemund完成验二维DNA折纸结构, Douglas等人在2009年固性三维DNA折纸术。同时,Jørgen Kjems的实验室用二维平面制成了三维结构。
DNA纳米科技最初受到一些质疑,因为核酸被当作非生物的材料建造结构和进行计算,并且远离实际应用。西曼1991年关于DNA立方体分析的论文被科学杂志拒绝,在此之前,一个评论家称赞他的独创性而另一个批评论文与生物学相关性不大。然而,2010年代,这个领域在基础科学方面研究的应用开始被认识到,并且其在医学和其他领域的应用被认为是可行的。2001年还只有很少的实验室做这方面研究,到2010年至少有60个实验室,也提高了人才储备量。因此在这十年内这方面的研究取得了很大进步。
參見
参考资料
- ^ 1.0 1.1 DNA polyhedra: Goodman, Russel P.; Schaap, Iwan A. T.; Tardin, C. F.; Erben, Christof M.; Berry, Richard M.; Schmidt, C.F.; Turberfield, Andrew J. Rapid chiral assembly of rigid DNA building blocks for molecular nanofabrication. Science. 9 December 2005, 310 (5754): 1661–1665. Bibcode:2005Sci...310.1661G. PMID 16339440. doi:10.1126/science.1120367.
- ^ RNA nanotechnology: Chworos, Arkadiusz; Severcan, Isil; Koyfman, Alexey Y.; Weinkam, Patrick; Oroudjev, Emin; Hansma, Helen G.; Jaeger, Luc. Building Programmable Jigsaw Puzzles with RNA. Science. 2004, 306 (5704): 2068–2072. Bibcode:2004Sci...306.2068C. PMID 15604402. doi:10.1126/science.1104686.
- ^ RNA nanotechnology: Guo, Peixuan. The Emerging Field of RNA Nanotechnology. Nature Nanotechnology. 2010, 5 (12): 833–842. Bibcode:2010NatNa...5..833G. PMC 3149862
 . PMID 21102465. doi:10.1038/nnano.2010.231.
. PMID 21102465. doi:10.1038/nnano.2010.231.
- ^ 4.0 4.1 4.2 Overview: Mao, Chengde. The emergence of complexity: lessons from DNA. PLoS Biology. 2004, 2 (12): 2036–2038. PMC 535573
 . PMID 15597116. doi:10.1371/journal.pbio.0020431. 已忽略未知参数
. PMID 15597116. doi:10.1371/journal.pbio.0020431. 已忽略未知参数|month=(建议使用|date=) (帮助) - ^ 5.0 5.1 5.2 引用错误:没有为名为
Seeman-sciam的参考文献提供内容 - ^ Background: Pelesko, John A. Self-assembly: the science of things that put themselves together. New York: Chapman & Hall/CRC. 2007: 5, 7. ISBN 978-1-58488-687-7.
- ^ 7.0 7.1 7.2 7.3 引用错误:没有为名为
Seeman2010的参考文献提供内容 - ^ Background: Long, Eric C. Fundamentals of nucleic acids. Hecht, Sidney M (编). Bioorganic chemistry: nucleic acids. New York: Oxford University Press. 1996: 4–10. ISBN 0-19-508467-5.
- ^ 9.0 9.1 9.2 9.3 Dynamic DNA nanotechnology: doi:10.1038/nchem.957
{{cite doi}}已停用,请参见{{cite journal}}。 - ^ 10.0 10.1 10.2 10.3 10.4 Structural DNA nanotechnology: Seeman, Nadrian C. An overview of structural DNA nanotechnology. Molecular Biotechnology. 2007, 37 (3): 246–257. PMID 17952671. doi:10.1007/s12033-007-0059-4. 已忽略未知参数
|month=(建议使用|date=) (帮助) - ^ Dynamic DNA nanotechnology: doi:10.1016/j.copbio.2006.10.004
{{cite doi}}已停用,请参见{{cite journal}}。 - ^ 12.0 12.1 12.2 12.3 Design: doi:10.1002/anie.200502358
{{cite doi}}已停用,请参见{{cite journal}}。 - ^ 13.0 13.1 Design: Brenneman, Arwen; Condon, Anne. Strand design for biomolecular computation. Theoretical Computer Science. 25 September 2002, 287: 39–58. doi:10.1016/S0304-3975(02)00135-4.
- ^ Overview: Lin, Chenxiang; Liu, Yan; Rinker, Sherri; Yan, Hao. DNA tile based self-assembly: building complex nanoarchitectures. ChemPhysChem. 11 August 2006, 7 (8): 1641–1647. PMID 16832805. doi:10.1002/cphc.200600260.
- ^ 15.0 15.1 15.2 15.3 15.4 15.5 15.6 15.7 Overview: doi:10.1038/nnano.2011.187
{{cite doi}}已停用,请参见{{cite journal}}。 - ^ 16.0 16.1 DNA origami: Rothemund, Paul W. K. Folding DNA to create nanoscale shapes and patterns. Nature. 16 March 2006, 440 (7082): 297–302. Bibcode:2006Natur.440..297R. PMID 16541064. doi:10.1038/nature04586.
- ^ 17.0 17.1 17.2 Kinetic assembly: Yin, Peng; Choi, Harry M. T.; Calvert, Colby R.; Pierce, Niles A. Programming biomolecular self-assembly pathways. Nature. 17 January 2008, 451 (7176): 318–322. Bibcode:2008Natur.451..318Y. PMID 18202654. doi:10.1038/nature06451.
- ^ 18.0 18.1 Design: Dirks, Robert M.; Lin, Milo; Winfree, Erik & Pierce, Niles A. Paradigms for computational nucleic acid design. Nucleic Acids Research. 15 February 2004, 32 (4): 1392–1403. PMC 390280
 . PMID 14990744. doi:10.1093/nar/gkh291.
. PMID 14990744. doi:10.1093/nar/gkh291.
- ^ Other arrays: Strong, Michael. Protein Nanomachines. PLoS Biology. 2004, 2 (3): e73. PMC 368168
 . PMID 15024422. doi:10.1371/journal.pbio.0020073. 已忽略未知参数
. PMID 15024422. doi:10.1371/journal.pbio.0020073. 已忽略未知参数|month=(建议使用|date=) (帮助) - ^ doi:10.1126/science.1089389
{{cite doi}}已停用,请参见{{cite journal}}。 - ^ 21.0 21.1 Algorithmic self-assembly: Rothemund, Paul W. K.; Papadakis, Nick; Winfree, Erik. Algorithmic self-assembly of DNA Sierpinski triangles. PLoS Biology. 2004, 2 (12): 2041–2053. PMC 534809
 . PMID 15583715. doi:10.1371/journal.pbio.0020424. 已忽略未知参数
. PMID 15583715. doi:10.1371/journal.pbio.0020424. 已忽略未知参数|month=(建议使用|date=) (帮助); - ^ DX arrays: Winfree, Erik; Liu, Furong; Wenzler, Lisa A.; Seeman, Nadrian C. Design and self-assembly of two-dimensional DNA crystals. Nature. 6 August 1998, 394 (6693): 529–544. Bibcode:1998Natur.394..539W. PMID 9707114. doi:10.1038/28998.
- ^ DX arrays: Liu, Furong; Sha, Ruojie; Seeman, Nadrian C. Modifying the surface features of two-dimensional DNA crystals. Journal of the American Chemical Society. 10 February 1999, 121 (5): 917–922. doi:10.1021/ja982824a.
- ^ Other arrays: Constantinou, Pamela E.; Wang, Tong; Kopatsch, Jens; Israel, Lisa B.; Zhang, Xiaoping; Ding, Baoquan; Sherman, William B.; Wang, Xing; Zheng, Jianping; Sha, Ruojie; Seeman, Nadrian C. Double cohesion in structural DNA nanotechnology. Organic and Biomolecular Chemistry. 21 September 2006, 4 (18): 3414–3419. PMID 17036134. doi:10.1039/b605212f.
- ^ Other arrays: Mathieu, Frederick; Liao, Shiping; Kopatsch, Jens; Wang, Tong; Mao, Chengde; Seeman, Nadrian C. Six-helix bundles designed from DNA. Nano Letters. 2005, 5 (4): 661–665. Bibcode:2005NanoL...5..661M. PMID 15826105. doi:10.1021/nl050084f. 已忽略未知参数
|month=(建议使用|date=) (帮助); - ^ 26.0 26.1 引用错误:没有为名为
SeemanGrowing的参考文献提供内容 - ^ Algorithmic self-assembly: Barish, Robert D.; Rothemund, Paul W. K.; Winfree, Erik. Two computational primitives for algorithmic self-assembly: copying and counting. Nano Letters. 2005, 5 (12): 2586–2592. Bibcode:2005NanoL...5.2586B. PMID 16351220. doi:10.1021/nl052038l. 已忽略未知参数
|month=(建议使用|date=) (帮助) - ^ DNA nanotubes: Rothemund, Paul W. K.; Ekani-Nkodo, Axel; Papadakis, Nick; Kumar, Ashish; Fygenson, Deborah Kuchnir & Winfree, Erik. Design and Characterization of Programmable DNA Nanotubes. Journal of the American Chemical Society. 22 December 2004, 126 (50): 16344–16352. PMID 15600335. doi:10.1021/ja044319l.
- ^ DNA nanotubes: Yin, P.; Hariadi, R. F.; Sahu, S.; Choi, H. M. T.; Park, S. H.; Labean, T. H.; Reif, J. H. Programming DNA Tube Circumferences. Science. 8 August 2008, 321 (5890): 824–826. Bibcode:2008Sci...321..824Y. PMID 18687961. doi:10.1126/science.1157312.
- ^ Three-dimensional arrays: Zheng, Jianping; Birktoft, Jens J.; Chen, Yi; Wang, Tong; Sha, Ruojie; Constantinou, Pamela E.; Ginell, Stephan L.; Mao, Chengde; Seeman, Nadrian C. From molecular to macroscopic via the rational design of a self-assembled 3D DNA crystal. Nature. 3 September 2009, 461 (7260): 74–77. Bibcode:2009Natur.461...74Z. PMC 2764300
 . PMID 19727196. doi:10.1038/nature08274.
. PMID 19727196. doi:10.1038/nature08274.
- ^ DNA polyhedra: Zhang, Yuwen; Seeman, Nadrian C. Construction of a DNA-truncated octahedron. Journal of the American Chemical Society. 1 March 1994, 116 (5): 1661–1669. doi:10.1021/ja00084a006.
- ^ DNA polyhedra: Shih, William M.; Quispe, Joel D.; Joyce, Gerald F. A 1.7-kilobase single-stranded DNA that folds into a nanoscale octahedron. Nature. 12 February 2004, 427 (6975): 618–621. PMID 14961116. doi:10.1038/nature02307.
- ^ DNA origami: Douglas, Shawn M.; Dietz, Hendrik; Liedl, Tim; Högberg, Björn; Graf, Franziska; Shih, William M. Self-assembly of DNA into nanoscale three-dimensional shapes. Nature. 21 May 2009, 459 (7245): 414–418. Bibcode:2009Natur.459..414D. PMC 2688462
 . PMID 19458720. doi:10.1038/nature08016.
. PMID 19458720. doi:10.1038/nature08016.
- ^ 34.0 34.1 DNA boxes: Andersen, Ebbe S.; Dong, Mingdong; Nielsen, Morten M.; Jahn, Kasper; Subramani, Ramesh; Mamdouh, Wael; Golas, Monika M.; Sander, Bjoern; Stark, Holger. Self-assembly of a nanoscale DNA box with a controllable lid. Nature. 7 May 2009, 459 (7243): 73–76. Bibcode:2009Natur.459...73A. PMID 19424153. doi:10.1038/nature07971.
- ^ DNA boxes: Ke, Yonggang; Sharma, Jaswinder; Liu, Minghui; Jahn, Kasper; Liu, Yan; Yan, Hao. Scaffolded DNA origami of a DNA tetrahedron molecular container. Nano Letters. 10 June 2009, 9 (6): 2445–2447. Bibcode:2009NanoL...9.2445K. PMID 19419184. doi:10.1021/nl901165f.
- ^ Overview: doi:10.1002/cbic.200900286
{{cite doi}}已停用,请参见{{cite journal}}。 - ^ Nanoarchitecture: Zheng, Jiwen; Constantinou, Pamela E.; Micheel, Christine; Alivisatos, A. Paul; Kiehl, Richard A.; Seeman Nadrian C. 2D Nanoparticle Arrays Show the Organizational Power of Robust DNA Motifs. Nano Letters. 2006, 6 (7): 1502–1504. Bibcode:2006NanoL...6.1502Z. PMID 16834438. doi:10.1021/nl060994c. 已忽略未知参数
|month=(建议使用|date=) (帮助); - ^ Nanoarchitecture: Park, Sung Ha; Pistol, Constantin; Ahn, Sang Jung; Reif, John H.; Lebeck, Alvin R.; Dwyer, Chris; LaBean, Thomas H. Finite-size, fully addressable DNA tile lattices formed by hierarchical assembly procedures. Angewandte Chemie. 2006, 118 (40): 749–753. doi:10.1002/ange.200690141. 已忽略未知参数
|month=(建议使用|date=) (帮助) - ^ Nanoarchitecture: Cohen, Justin D.; Sadowski, John P.; Dervan, Peter B. Addressing single molecules on DNA nanostructures. Angewandte Chemie International Edition. 22 October 2007, 46 (42): 7956–7959. PMID 17763481. doi:10.1002/anie.200702767.
- ^ Nanoarchitecture: Maune, Hareem T.; Han, Si-Ping; Barish, Robert D.; Bockrath, Marc; Goddard, III, William A.; Rothemund, Paul W. K.; Winfree, Erik. Self-assembly of carbon nanotubes into two-dimensional geometries using DNA origami templates. Nature Nanotechnology. 2009, 5 (1): 61–66. Bibcode:2010NatNa...5...61M. PMID 19898497. doi:10.1038/nnano.2009.311. 已忽略未知参数
|month=(建议使用|date=) (帮助) - ^ Nanoarchitecture: doi:10.1021/nn1035075
{{cite doi}}已停用,请参见{{cite journal}}。 - ^ Nanoarchitecture: doi:10.1002/anie.200460257
{{cite doi}}已停用,请参见{{cite journal}}。 - ^ 43.0 43.1 43.2 引用错误:没有为名为
nanomachines的参考文献提供内容 - ^ DNA machines: Mao, Chengde; Sun, Weiqiong; Shen, Zhiyong; Seeman, Nadrian C. A DNA nanomechanical device based on the B-Z transition. Nature. 14 January 1999, 397 (6715): 144–146. Bibcode:1999Natur.397..144M. PMID 9923675. doi:10.1038/16437.
- ^ DNA machines: Yurke, Bernard; Turberfield, Andrew J.; Mills, Allen P., Jr; Simmel, Friedrich C.; Neumann, Jennifer L. A DNA-fuelled molecular machine made of DNA. Nature. 10 August 2000, 406 (6796): 605–609. Bibcode:2000Natur.406..605Y. PMID 10949296. doi:10.1038/35020524.
- ^ DNA machines: Yan, Hao; Zhang, Xiaoping; Shen, Zhiyong; Seeman, Nadrian C. A robust DNA mechanical device controlled by hybridization topology. Nature. 3 January 2002, 415 (6867): 62–65. Bibcode:2002Natur.415...62Y. PMID 11780115. doi:10.1038/415062a.
- ^ DNA machines: doi:10.1002/ange.200351818
{{cite doi}}已停用,请参见{{cite journal}}。 - ^ DNA machines: doi:10.1038/nnano.2008.3
{{cite doi}}已停用,请参见{{cite journal}}。 - ^ Applications: doi:10.1126/science.1214081
{{cite doi}}已停用,请参见{{cite journal}}。 - ^ DNA walkers: Shin, Jong-Shik; Pierce, Niles A. A synthetic DNA walker for molecular transport. Journal of the American Chemical Society. 8 September 2004, 126 (35): 10834–10835. PMID 15339155. doi:10.1021/ja047543j.
- ^ DNA walkers: Sherman, William B.; Seeman, Nadrian C. A precisely controlled DNA biped walking device. Nano Letters. 2004, 4 (7): 1203–1207. Bibcode:2004NanoL...4.1203S. doi:10.1021/nl049527q. 已忽略未知参数
|month=(建议使用|date=) (帮助) - ^ DNA walkers: Tian, Ye; He, Yu; Chen, Yi; Yin, Peng; Mao, Chengde. A DNAzyme that walks processively and autonomously along a one-dimensional track. Angewandte Chemie. 11 July 2005, 117 (28): 4429–4432. doi:10.1002/ange.200500703.
- ^ DNA walkers: Bath, Jonathan; Green, Simon J.; Turberfield, Andrew J. A free-running DNA motor powered by a nicking enzyme. Angewandte Chemie International Edition. 11 July 2005, 44 (28): 4358–4361. doi:10.1002/anie.200501262.
- ^ Functional DNA walkers: Lund, Kyle; Manzo, Anthony J.; Dabby, Nadine; Michelotti, Nicole; Johnson-Buck, Alexander; Nangreave, Jeanette; Taylor, Steven; Pei, Renjun; Stojanovic, Milan N.; Walter Nils G.; Winfree, Erik; Yan, Hao. Molecular robots guided by prescriptive landscapes. Nature. 13 May 2010, 465 (7295): 206–210. Bibcode:2010Natur.465..206L. PMC 2907518
 . PMID 20463735. doi:10.1038/nature09012.
. PMID 20463735. doi:10.1038/nature09012.
- ^ Functional DNA walkers: He, Yu; Liu, David R. Autonomous multistep organic synthesis in a single isothermal solution mediated by a DNA walker. Nature Nanotechnology. 2010, 5 (11): 778–782. Bibcode:2010NatNa...5..778H. PMC 2974042
 . PMID 20935654. doi:10.1038/nnano.2010.190. 已忽略未知参数
. PMID 20935654. doi:10.1038/nnano.2010.190. 已忽略未知参数|month=(建议使用|date=) (帮助) - ^ Strand displacement cascades: doi:10.1126/science.1132493
{{cite doi}}已停用,请参见{{cite journal}}。 - ^ Strand displacement cascades: doi:10.1126/science.1200520
{{cite doi}}已停用,请参见{{cite journal}}。 - ^ 58.0 58.1 引用错误:没有为名为
growsup的参考文献提供内容 - ^ Applications: Rietman, Edward A. Molecular engineering of nanosystems. Springer. 2001: 209–212 [17 April 2011]. ISBN 978-0-387-98988-4.
- ^ Applications: Jungmann, Ralf; Renner, Stephan; Simmel, Friedrich C. From DNA nanotechnology to synthetic biology. HFSP journal. 2008, 2 (2): 99–109. PMC 2645571
 . PMID 19404476. doi:10.2976/1.2896331. 已忽略未知参数
. PMID 19404476. doi:10.2976/1.2896331. 已忽略未知参数|month=(建议使用|date=) (帮助)
扩展阅读
General:
- Seeman, Nadrian C. Nanotechnology and the double helix. Scientific American. 2004, 290 (6): 64–75. PMID 15195395. doi:10.1038/scientificamerican0604-64. 已忽略未知参数
|month=(建议使用|date=) (帮助)—An article written for laypeople by the founder of the field - Seeman, Nadrian C. Structural DNA nanotechnology: growing along with Nano Letters. Nano Letters. 9 June 2010, 10 (6): 1971–1978. Bibcode:2010NanoL..10.1971S. PMC 2901229
 . PMID 20486672. doi:10.1021/nl101262u.—A review of results in the period 2001–2010
. PMID 20486672. doi:10.1021/nl101262u.—A review of results in the period 2001–2010 - Seeman, Nadrian C. Nanomaterials based on DNA. Annual Review of Biochemistry. 2010, 79: 65–87. PMID 20222824. doi:10.1146/annurev-biochem-060308-102244.—A more comprehensive review including both old and new results in the field
- Service, Robert F. DNA nanotechnology grows up. Science. 3 June 2011, 332 (6034): 1140–1143. doi:10.1126/science.332.6034.1140. and doi:10.1126/science.332.6034.1142.—A news article focusing on the history of the field and development of new applications
- Zadegan, Reza M.; Norton, Michael L. Structural DNA Nanotechnology: From Design to Applications. Int. J. Mol. Sci. 2012, 13 (6): 7149–7162. PMC 3397516
 . PMID 22837684. doi:10.3390/ijms13067149. 已忽略未知参数
. PMID 22837684. doi:10.3390/ijms13067149. 已忽略未知参数|month=(建议使用|date=) (帮助)—A very recent and comprehensive review in the field
Specific subfields:
- Bath, Jonathan; Turberfield, Andrew J. DNA nanomachines. Nature Nanotechnology. 5 May 2007, 2 (5): 275–284. Bibcode:2007NatNa...2..275B. PMID 18654284. doi:10.1038/nnano.2007.104.—A review of nucleic acid nanomechanical devices
- Feldkamp, Udo; Niemeyer, Christof M. Rational design of DNA nanoarchitectures. Angewandte Chemie International Edition. 13 March 2006, 45 (12): 1856–76. PMID 16470892. doi:10.1002/anie.200502358.—A review coming from the viewpoint of secondary structure design
- Lin, Chenxiang; Liu, Yan; Rinker, Sherri; Yan, Hao. DNA tile based self-assembly: building complex nanoarchitectures. ChemPhysChem. 11 August 2006, 7 (8): 1641–1647. PMID 16832805. doi:10.1002/cphc.200600260.—A minireview specifically focusing on tile-based assembly
- Zhang, David Yu; Seelig, Georg. Dynamic DNA nanotechnology using strand-displacement reactions. Nature Chemistry. 2011, 3 (2): 103–113. Bibcode:2011NatCh...3..103Z. PMID 21258382. doi:10.1038/nchem.957. 已忽略未知参数
|month=(建议使用|date=) (帮助)—A review of DNA systems making use of strand displacement mechanisms
外部链接
- International Society for Nanoscale Science, Computation and Engineering
- What is Bionanotechnology?—a video introduction to DNA nanotechnology




