組織蛋白

組織蛋白(英語:histone,或稱組蛋白)是真核生物體細胞染色質與原核細胞中的鹼性蛋白質,和DNA共同組成核小體結構[1][2][3]。它們是染色質的主要蛋白質組分,作為DNA纏繞的線軸,並在基因調控中發揮作用,但是原核細胞組蛋白對基因調控的作用非常微弱[3]。沒有組織蛋白,染色體中未纏繞的DNA將非常長(人類DNA中的長寬比超過1000萬比1)。例如,每個人類二倍體細胞(含有23對染色體)具有約1.8米長的DNA,但是在組織蛋白上纏繞它具有大約90微米(0.09毫米)的染色質,當在有絲分裂期間複製和濃縮時,其導致約120微米的染色體[4]。
| 核心組織蛋白H2A/H2B/H3/H4 | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
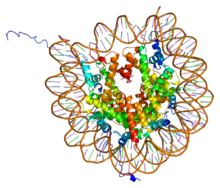 PDB rendering of Complex between nucleosome core particle (h3,h4,h2a,h2b) and 146 bp long DNA fragment based on 1aoi. | |||||||||||
| 鑑定 | |||||||||||
| 標誌 | Histone | ||||||||||
| Pfam | PF00125(舊版) | ||||||||||
| Pfam宗系 | CL0012(舊版) | ||||||||||
| InterPro | IPR007125 | ||||||||||
| SCOP | 1hio / SUPFAM | ||||||||||
| |||||||||||
| 連接組織蛋白H1和H5家族 | |||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
 | |||||||||||
| 鑑定 | |||||||||||
| 標誌 | Linker_histone | ||||||||||
| Pfam | PF00538(舊版) | ||||||||||
| InterPro | IPR005818 | ||||||||||
| SMART | SM00526 | ||||||||||
| SCOP | 1hst / SUPFAM | ||||||||||
| |||||||||||
分類和組織蛋白變體
[編輯]存在五個主要的組織蛋白家族: H1/H5,H2A,H2B,H3和H4[2][5][6][7] 。組織蛋白H2A,H2B,H3和H4被稱為核心組織蛋白(Core Histone),而組織蛋白H1/H5被稱為連接組織蛋白(Linker Histone)。
在H2A、H2B、H3及H4這四種組織蛋白中的其中兩種稱為「核心組織蛋白」,並且集合成為八聚體的核小體核心顆粒。這種集合是將DNA的146對鹼基對以1.65左手超螺旋形圍在這個蛋白質線軸。連接組織蛋白H1將核小體核心顆粒與DNA的進入位點及E位點結合,因而可以將DNA緊扣在位,並且能容許形成更高層次的結構。最基本的形狀為一個10納米的纖維或一連串的珠子。這涉及將在每一個核小體之間約50對的DNA鹼基對圍在這些核小體上,這些DNA又稱為連接DNA。較高層次的結構包括有30及100納米的纖維,是在一般細胞內的結構。在減數分裂中,透過核小體與其他蛋白質的相互作用,合成染色體。合成的組織蛋白與DNA稱為染色質。
核心組織蛋白是高度保守的蛋白質,意即組織蛋白在氨基酸序列中有著非常小的改變。連接組織蛋白通常有著多於一種的形狀,對比核心組織蛋白是保守程度較低的。
在主要的組織蛋白類別中,亦存在一些異構體。它們有著相同的氨基酸序列及相似的核心結構,但卻有著不同的特徵。這些不同的組織蛋白通常帶著染色質的特別功能,就如與H3相似的CenpA是唯一的組織蛋白與染色體的著絲點聯合;H2A的異構體H2A.Z是與活性轉錄基因聯合與涉及在異染色體的形成;另一個H2A異構體H2A.X以雙鏈斷裂與DNA結合,並進行DNA修復。
H1
[編輯]組織蛋白H1有著一個中央球狀結構域及長的C與N端尾巴,能將小珠子串結構圍成30納米大小的螺線管結構。對比其他種類的組織蛋白,H1的數量只為其他的一半。這是因為它不是構成核小體部份,而只是將DNA及核小體緊扣在一起。H1亦有著它的異構體,稱為組織蛋白H5。
H2A、H2B及H4
[編輯]組織蛋白H2A、H2B及H4同樣有著一個主要的球狀結構域與長的N端尾巴,是組成小珠子串結構內的核小體的重要單元。
H3
[編輯]與組織蛋白H2A及H2B類似,組織蛋白H3有著一個主要的球狀結構域與長的N端尾巴,是組成小珠子串結構內的核小體的重要原素。它的N端尾巴從球狀核小體核心伸出,能進行多種影響細胞運作的表觀遺傳修飾。這些修飾包括將甲基及乙酰基共價附著於離氨酸或精氨酸,及絲氨酸或羥丁氨酸的磷酸化。離氨酸9的甲基化涉及基因沉默及在DNA內形成相對較不活躍的異染色質。組織蛋白H3的乙酰基化會在組織蛋白尾巴內不同的離氨酸位置出現,並且由組織蛋白乙酰轉移酶(HAT)所催化。離氨酸14的乙酰基化在基因中很普遍,亦會轉錄成為核糖核酸(RNA)。
以下是人類組蛋白的列表:
| 超級家族 | 家族 | 亞家族 | 成員 |
|---|---|---|---|
| 連接組織蛋白 | H1 | H1F | H1F0, H1FNT, H1FOO, H1FX |
| H1H1 | HIST1H1A, HIST1H1B, HIST1H1C, HIST1H1D, HIST1H1E, HIST1H1T | ||
| 核心組織蛋白 | H2A | H2AF | H2AFB1, H2AFB2, H2AFB3, H2AFJ, H2AFV, H2AFX, H2AFY, H2AFY2, H2AFZ |
| H2A1 | HIST1H2AA, HIST1H2AB, HIST1H2AC, HIST1H2AD, HIST1H2AE, HIST1H2AG, HIST1H2AI, HIST1H2AJ, HIST1H2AK, HIST1H2AL, HIST1H2AM | ||
| H2A2 | HIST2H2AA3, HIST2H2AC | ||
| H2B | H2BF | H2BFM, H2BFS, H2BFWT | |
| H2B1 | HIST1H2BA, HIST1H2BB, HIST1H2BC, HIST1H2BD, HIST1H2BE, HIST1H2BF, HIST1H2BG, HIST1H2BH, HIST1H2BI, HIST1H2BJ, HIST1H2BK, HIST1H2BL, HIST1H2BM, HIST1H2BN, HIST1H2BO | ||
| H2B2 | HIST2H2BE | ||
| H3 | H3A1 | HIST1H3A, HIST1H3B, HIST1H3C, HIST1H3D, HIST1H3E, HIST1H3F, HIST1H3G, HIST1H3H, HIST1H3I, HIST1H3J | |
| H3A2 | HIST2H3C | ||
| H3A3 | HIST3H3 | ||
| H4 | H41 | HIST1H4A, HIST1H4B, HIST1H4C, HIST1H4D, HIST1H4E, HIST1H4F, HIST1H4G, HIST1H4H, HIST1H4I, HIST1H4J, HIST1H4K, HIST1H4L | |
| H44 | HIST4H4 |
結構
[編輯]核小體核心顆粒是由兩個H2A-H2B二聚物及兩個H3-H4二聚物結合而成,形成兩半接近對稱的蛋白質三級結構(2階旋轉對稱,即1個高分子是另一個的鏡像)。H2A-H2B及H3-H4二聚物本身亦呈現偽雙向對稱。
這四個核心組織蛋白(H2A、H2B、H3及H4)是在結構上相似及在進化中高度保存的,所有均有著一個「螺旋纏繞螺旋纏繞螺旋」的形狀,可以容許簡單的二聚化。它們在胺基酸結構上都有著一條長的尾巴,讓轉錄後修飾的進行。
總括來說,組織蛋白與DNA有著五種的相互作用:
- H2B、H3及H4的α螺旋兩極積聚正電,能與DNA的帶有負電荷的磷酸鹽分子團產生相互作用。
- 在DNA骨幹與胺基之間的氫鍵對組織蛋白的主鏈。
- 組織蛋白與DNA的脫氧核糖的非極性相互作用。
- 鹼性胺基酸(如離氨酸及精氨酸)旁鏈與DNA磷酸氧旁鏈之間的鹽連及氫鍵。
- H3及H2B的N端尾巴的非特定副槽面插入至DNA分子的兩個副槽面。
組織蛋白最重要的基本性質,除了是協助與DNA的相互作用外,就是它的水溶性。
組織蛋白的轉譯後修飾會先在它的N端尾巴開始,再而在其球狀結構域進行。這種修飾包括有甲基化、瓜氨酸化、乙醯化、磷酸化、小泛素相關修飾化、泛素化及二磷酸腺苷核糖基化。這些影響著組織蛋白在基因調控的功能。
一般來說,活性的基因較少與組織蛋白聯繫,但非活性的基因會在間期中與組織蛋白聯合。組織蛋白的結構在進化上保存,這是因為任何有害的突變會造成嚴重的不適應性。
歷史
[編輯]於1884年,艾布瑞契·科塞爾首先發現組織蛋白。直至1990年代早期,組織蛋白才被更多認識,並非純粹細胞核的惰性填充料,這部分基於馬克·普塔什尼(Mark Ptashne)等人的模型,他們認為轉錄是被蛋白質-DNA和蛋白質-蛋白質相互作用在很大程度上被激活裸DNA模板,就像細菌一樣。及後它的調控功能才被發現。
在1980年代,Yahli Lorch和羅傑·科恩伯格(Roger Kornberg)[8]表明,核心啟動子上的核小體體外阻止了轉錄的啟動,邁克爾·格倫斯坦(Michael Grunstein)[9]證明組蛋白在體內抑制轉錄,導致核小體為 一般基因阻遏物。
功能
[編輯]填充蛋白質
[編輯]組織蛋白作為DNA纏繞的線軸。這使得能夠在細胞核內將真核細胞的大型的基因組所必需的壓實物:壓實的分子比未壓實的分子短40,000倍。
染色質調控
[編輯]組織蛋白進行翻譯後修飾,以更改它與DNA及其他核蛋白的相互作用。組織蛋白H3及H4有着核小體伸出的長尾巴,能夠在不同的地方進行共價修飾。這種修飾包括有甲基化、瓜氨化、乙酰基化、磷酸化、小泛素相關修飾化、泛素化及二磷酸腺苷核糖基化。組織蛋白核心(即H2A及H3)亦可以作出修飾。修飾的組合可以組成編碼,成為組織蛋白編碼。組織蛋白修飾在不同的生物過程起着作用,包括基因表觀調控、DNA修復、有絲分裂及減數分裂 [10]。
組織蛋白修飾的命名是:
舉例來說,H3K4Me就代表組織蛋白H3從N端開始起計第4個賴氨酸的甲基化。
參考文獻
[編輯]- ^ Youngson RM. Collins Dictionary of Human Biology. Glasgow: HarperCollins. 2006. ISBN 0-00-722134-7.
- ^ 2.0 2.1 Cox M, Nelson DR, Lehninger AL. Lehninger Principles of Biochemistry. San Francisco: W.H. Freeman. 2005. ISBN 0-7167-4339-6.
- ^ 3.0 3.1 楊榮武, 生物化學原理第三版. 基因表达调控. 北京: 高等教育出版社. 2018. ISBN 9787040500813.
- ^ Redon C, Pilch D, Rogakou E, Sedelnikova O, Newrock K, Bonner W. Histone H2A variants H2AX and H2AZ. Current Opinion in Genetics & Development. Apr 2002, 12 (2): 162–9. PMID 11893489. doi:10.1016/S0959-437X(02)00282-4.
- ^ Histone Variants Database 2.0. National Center for Biotechnology Information. [13 January 2017]. (原始內容存檔於2016-12-20).
- ^ Bhasin M, Reinherz EL, Reche PA. Recognition and classification of histones using support vector machine. Journal of Computational Biology. 2006, 13 (1): 102–12. PMID 16472024. doi:10.1089/cmb.2006.13.102.
- ^ Hartl DL, Freifelder D, Snyder LA. Basic Genetics. Boston: Jones and Bartlett Publishers. 1988. ISBN 0-86720-090-1.
- ^ Lorch Y, LaPointe JW, Kornberg RD. Nucleosomes inhibit the initiation of transcription but allow chain elongation with the displacement of histones. Cell. Apr 1987, 49 (2): 203–10. PMID 3568125. doi:10.1016/0092-8674(87)90561-7.
- ^ Kayne PS, Kim UJ, Han M, Mullen JR, Yoshizaki F, Grunstein M. Extremely conserved histone H4 N terminus is dispensable for growth but essential for repressing the silent mating loci in yeast. Cell. Oct 1988, 55 (1): 27–39. PMID 3048701. doi:10.1016/0092-8674(88)90006-2.
- ^ Ning Song, Jie Liu, Shucai An, Tomoya Nishino, Yoshitaka Hishikawa and Takehiko Koji. Immunohistochemical analysis of histone H3 modifications in germ cells during mouse spermatogenesis. Acta Histochemica et Cytochemica. 2011, 44 (4). doi:10.1267/ahc.11027.[永久失效連結]
參見
[編輯]外部連結
[編輯]- HistoneDB 2.0 - 組織蛋白和變體數據庫 (頁面存檔備份,存於網際網路檔案館) -- 在美國國家生物技術信息中心(NCBI)
- Chromatin, Histones & Cathepsin (頁面存檔備份,存於網際網路檔案館); PMAP The Proteolysis Map-animation
