氧化磷酸化


氧化磷酸化(英語:oxidative phosphorylation,縮寫作 OXPHOS)是細胞的一種代謝途徑,該過程在真核生物的粒線體內膜或原核生物的細胞膜上發生,使用其中的酶及氧化各類營養素所釋放的能量來合成三磷酸腺苷(ATP)。雖然地球上的生物消耗的能源物質範圍極廣,為合成代謝直接提供能量的分子卻幾乎都是ATP。幾乎所有的好氧性生物都以三羧酸循環-氧化磷酸化作為製造ATP的主要過程。該途徑如此普遍的原因可能是:與其他的代謝途徑,特別是糖解之類的無氧發酵途徑相比,它能更高效地釋放能量。
氧化磷酸化期間,電子在氧化還原反應中從電子供體轉移到電子受體,例如氧。氧化還原反應所釋放的能量用於合成ATP。在真核生物中,這些氧化還原反應在一系列粒線體內膜上的蛋白質複合體的參與下完成,而在原核生物中,這些蛋白質存在於細胞膜間隙中。這一串蛋白質稱為電子傳遞鏈。真核生物包含五種主要的蛋白質複合體,而原核生物中存在許多不同的酶,以便利用各種電子供體和受體。
在「電子傳遞」過程中,質子被電子流過電子傳遞鏈所釋放的能量泵出粒線體內膜。這會以pH梯度和跨膜電位差的形式產生位能。儲存的能量通過讓質子順梯度跨膜內流,由稱為ATP合酶的大型酶所使用;這個過程稱為化學滲透。這種酶在磷酸化反應過程中就像一台機械馬達,酶的一部分在質子流的驅動下不停旋轉,將二磷酸腺苷(ADP)合成為三磷酸腺苷。
雖然氧化磷酸化是新陳代謝的重要組成部分,它卻會產生活性氧如超氧化物和過氧化氫,使自由基擴散開來,破壞細胞及造成病變,還有可能導致老化。該代謝途徑中的酶也是許多藥物和毒物所抑制的目標。
通過化學滲透轉移能量的概述
[編輯]氧化磷酸化的工作原理是利用釋放能量的化學反應來驅動需要能量的反應:這樣的反應稱為是偶聯反應。電子在電子傳遞鏈上從電子供體(如NADH)到電子受體(如氧)的流動,是一個放能的過程,而ATP的合成是一個耗能的過程,需要輸入能量。電子傳遞鏈和ATP合酶都在膜中,在稱為「化學滲透」的過程中,通過質子穿過這層膜的運動,將能量從電子傳遞鏈轉移到ATP合酶中[1]。實際上,這就像一個簡單的電路,質子通過電子傳遞鏈中的質子泵酶,從膜帶負電位的N端流向帶正電位的P端。這些酶如同其中的電池,作功來驅動電流在迴路中流動。質子的移動產生了跨膜電化學梯度,這通常稱為「質子動力」。它包含兩部分:質子濃度差(H+梯度,ΔpH)和電位差,其中N端有負電荷[2]。
ATP合酶接通電路,讓質子順著電化學梯度流動,回到膜的N端,從中釋放儲存的能量[3]。這動能驅動著酶的部分結構旋轉,並與ATP的合成相偶聯。
質子動力的兩種組分在熱力學上是等效的:在粒線體中,大部分能量由電位差提供;在嗜鹼性細菌中,電能甚至要補償pH逆差造成的反作用。與之相反,葉綠體主要依靠ΔpH工作。然而,因為ATP合成的動力學要求,它們也需要一個小的膜電位。至少在梭桿菌P. modestum的情況中,它驅動ATP合酶FO轉子的a、c亞基逆向旋轉[2]。
與通過厭氧發酵產生的能量相比較,通過氧化磷酸化釋放的能量很多。糖解只產生2個ATP分子,而通過將一分子葡萄糖轉變為二氧化碳和水而生成的10個NADH和兩分子琥珀酸氧化磷酸化,可以產生30到36個ATP[4]。以上的ATP產量均為理論最大值;在實際中,一些質子會發生跨膜洩漏,從而降低了ATP的產量[5]。
電子和質子的轉移分子
[編輯]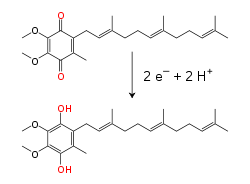
電子傳遞鏈能承載質子和電子,將電子從供體轉移到受體,並在膜間搬運質子。這些過程會使用可溶性的蛋白質結合轉移分子。在粒線體中,電子在膜間隙內由水溶性電子傳遞蛋白細胞色素c傳遞[6]。它通過其結構中血基質基團的一個鐵原子的還原和氧化過程傳輸電子。在某些細菌的周質空間中也發現了細胞色素c[7]。
粒線體內膜中的脂溶性電子載體輔酶Q10(Q)通過氧化還原循環,可同時攜帶電子和質子[8]。這個小苯醌分子疏水性很強,所以它能自由地在膜中擴散。當Q接受兩個電子和兩個質子時,它被還原為「泛酚」形式(QH2);當QH2釋放兩個電子和兩個質子時,它被氧化為「泛醌」(Q)形式。結果,如果安排兩種酶,使得Q在膜的一側被還原,而QH2在另一側被氧化,泛醌就能耦合這些反應,並攜帶質子穿過膜[9]。除了泛醌,一些細菌在電子傳遞鏈中還使用其他的醌類,如甲萘醌[10]。
在蛋白質中,電子在黃素輔因子[3][11]、鐵硫簇和細胞色素之間轉移。有幾種類型的鐵硫簇化合物。在電子傳遞鏈中發現的最簡單的一種包含了由兩個無機硫原子連接的兩個鐵原子;這些稱為[2Fe-2S]簇。第二種稱為[4Fe-4S],包含了由四個鐵原子和四個硫原子組成的立方體。在這些簇中,每個鐵原子與一個額外的胺基酸配位,通常是半胱胺酸中的硫原子。金屬離子輔因子在氧化還原反應中不結合或釋放質子,因此在電子傳遞鏈中都只通過蛋白質傳輸電子。電子沿著這些輔因子鏈跳躍,在蛋白質中移動很長的距離[12]。這些現象的本質是量子穿隧效應,在小於1.4×10−9米的距離內非常迅速[13]。
真核生物的電子傳遞鏈
[編輯]許多生化代謝過程,如糖解、三羧酸循環和β氧化,都會產生還原型輔酶NADH。此輔酶含有高電極電位的電子;也就是說,它們將在氧化時釋放出大量的能量。然而,細胞不會一次性釋放完全部的能量,因為在這種情況下,反應將無法控制。相反,電子從NADH釋放出來,並通過一系列的酶傳遞給氧氣,其中每步只釋放少量的能量。由複合體I到IV組成的這組酶稱為電子傳遞鏈,存在於粒線體內膜中。琥珀酸也被電子傳遞鏈氧化,但起點不同。
真核生物中,電子傳遞系統中的酶用從氧化NADH釋放的能量,泵送質子穿過粒線體內膜。這會使質子在膜間隙中積聚,產生跨膜電化學梯度。然後,ATP合酶使用存儲在其中的位能以產生ATP。其中,真核細胞粒線體中的氧化磷酸化是這一過程研究最為透徹的樣例。粒線體存在於幾乎所有的真核生物中,但部分原生生物例外,如陰道毛滴蟲中稱為氫化酶體的殘留粒線體會將質子還原為氫[14]。
| 呼吸酶 | 氧化還原對 | 中點電位(伏) |
|---|---|---|
| NADH去氫酶 | NAD+ / NADH | −0.32[15] |
| 琥珀酸去氫酶 | FMN或FAD / FMNH2或FADH2 | −0.20[15] |
| 細胞色素bc1複合體 | 輔酶Q10ox / 輔酶Q10red | +0.06[15] |
| 細胞色素bc1複合體 | 細胞色素box / 細胞色素bred | +0.12[15] |
| 複合體IV | 細胞色素cox / 細胞色素cred | +0.22[15] |
| 複合體IV | 細胞色素aox / 細胞色素ared | +0.29[15] |
| 複合體IV | O2 / HO− | +0.82[15] |
| 條件:pH = 7[15] | ||
NADH-輔酶Q氧化還原酶(複合體I)
[編輯]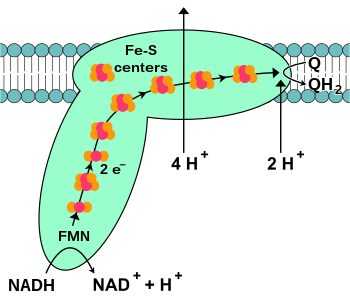
NADH-Q還原酶,又稱「NADH去氫酶」或「複合體I」,是電子傳遞鏈中的第一個蛋白[16]。它是一個巨大的酶,在哺乳動物中該複合體由46亞基組成,有1000千道爾頓(kDa)[17]。目前只了解一種細菌的複合體詳細結構[18][19];大多數生物的複合體酷似有一個大「球」的靴子,從膜中穿到粒線體[20][21]。編碼單個蛋白質的基因同時包含在細胞核和粒線體基因組中,許多粒線體酶都是這種情況。
此酶催化的反應為NADH被輔酶Q10,即粒線體膜上的一種脂溶性醌類「泛醌」(在下面公式中寫作Q)所氧化,同時轉移兩個電子:
這個反應乃至於整個電子鏈,是從NADH分子與複合體I結合及放出兩個電子而開始。電子通過附著在其上的黃素單核苷酸(FMN)輔基進入複合體I。電子的加入使FMN轉換為它的還原形式,FMNH2。電子隨後通過一系列存在於複合體中的第二種輔基鐵硫簇轉移[18]。複合體I中存在兩種鐵硫簇,[2Fe-2S]和[4Fe-4S]。
電子穿過複合體時,有4個質子從基質泵入膜間隙。尚不清楚其中的機制,但似乎與複合體I的構象改變有關,這可使該蛋白質在膜的N側結合質子,並在P側釋放它們[22]。最後,電子從鐵硫簇鏈轉移到膜中的泛醌分子上[16]。泛醌的還原也有助於質子梯度的產生,因為基質中的兩個質子被用於泛酚(QH2)的還原。
琥珀酸-Q氧化還原酶(複合體II)
[編輯]
琥珀酸-Q氧化還原酶,又稱「複合體II」或「琥珀酸去氫酶」,是電子傳遞鏈的第二個入口[23]。它很特別,因為它是唯一一個既屬於三羧酸循環、又屬於電子傳遞鏈的酶。複合體II包含四個蛋白質亞基,一個附著的黃素腺嘌呤二核苷酸(FAD)輔因子,鐵硫簇,和一個不參與將電子轉移到輔酶Q、但被認為在降低氧化物活性上起重要作用的血基質基團[24][25]。它將琥珀酸氧化為延胡索酸,將泛醌還原。該反應釋放的能量比氧化NADH少,因此複合體II不運輸質子穿過膜,不會影響質子梯度。
一些真核生物,如寄生蟲豬蛔蟲,有類似複合體II的延胡索酸還原酶(甲基萘醌:延胡索酸氧化還原酶,又稱QFR),但功能相反,其氧化泛醌而還原延胡索酸。這使蠕蟲可以在大腸的厭氧環境中生活,將延胡索酸作為電子受體進行厭氧氧化磷酸化[26]。複合體II的另一種非常規功能在引起瘧疾的寄生蟲「惡性瘧原蟲」中得到體現。在這裡作為氧化酶,複合體II的逆作用對泛酚的再生很重要,寄生蟲將其用於一個不尋常的生物合成嘧啶的方式[27]。
電子傳遞黃素蛋白-Q氧化還原酶
[編輯]電子傳遞黃素蛋白-泛醌氧化還原酶(ETF-Q氧化還原酶),又稱「電子傳遞-黃素蛋白去氫酶」,是電子傳遞鏈的第三個入口。它是接收粒線體基質中電子傳遞黃素蛋白的電子,並用這些電子還原泛醌的酶[28]。這種酶包含一個黃素和一個[4Fe-4S]簇,但不像其它的呼吸鏈複合體,它只附著在膜的表面,不跨越脂質雙分子層[29]。
哺乳動物中,該代謝途徑在脂肪酸的β氧化和胺基酸及膽鹼的分解代謝作用中很重要,因為它接受來自多個乙醯輔酶A去氫酶的電子[30][31]。在植物中,ETF-Q氧化還原酶也對使植物可以在長時間的黑暗中生存下來的代謝反應重要[32]。
Q-細胞色素c氧化還原酶(複合體III)
[編輯]
Q-細胞色素c氧化還原酶又稱「細胞色素c還原酶」、「細胞色素bc1複合體」,或簡稱「複合體III」[33][34]。在哺乳動物中,這種酶是一個二聚體,每個亞基包含11個蛋白質亞基,1個[2Fe-2S]鐵硫簇和3個細胞色素:1個細胞色素c1和2個細胞色素b[35]。細胞色素是一種傳輸電子的蛋白,包含至少一個血基質基團。當電子通過蛋白傳遞時,複合體III中血基質基團內的鐵原子在還原型二價鐵(+2)和氧化型三價鐵(+3)之間切換。
複合體III催化的反應是一分子泛酚的氧化和兩分子細胞色素c的還原,血基質蛋白部分與粒線體相關。不同於輔酶Q攜帶兩個電子,細胞色素c只攜帶一個電子。
作為反應中唯一可將電子從QH2供體轉移到細胞色素c受體的物質,複合體Ⅲ的反應機理較其他呼吸複合物認識更為詳盡,其包含兩個步驟,稱作Q循環[36]。首先在第一步中,酶與三個QH2受質結合,然後將其氧化,並將所產生的一個電子傳遞到第二個受質細胞色素C。QH2所釋放的兩個質子被傳遞到膜間隙。第三個受質Q接受來自QH2的第二個電子,而還原為泛半醌自由基Q.-。先前的兩個受質被釋放,但這個泛半醌中間體仍然被酶所結合。第二步中,第二個QH2分子與酶結合,再一次將它的第一個電子傳遞給細胞色素c受體。結合的泛半醌得到第二個電子,並獲得粒線體基質中的兩個質子,而還原為QH2。這個QH2隨後被酶釋放[37]。
當輔酶Q在膜的內側還原為泛酚,而在另一面氧化為泛醌時,質子的跨膜淨轉移隨之發生,從而增加了質子梯度[3]。通過相當複雜的兩步機制完成反應是重要的,因為它增加了質子轉移的效率。如果替換Q循環,直接用一個QH2分子來還原兩個細胞色素c分子,則每還原一個細胞色素c只能傳送一個質子,效率將會減半[3]。
細胞色素c氧化酶(複合體IV)
[編輯]
細胞色素c氧化酶,又稱「複合體IV」,是在電子傳遞鏈的最後一個蛋白質複合體[38]。哺乳動物的酶有極其複雜的結構,包含13個亞基,2個血基質基團,以及多種金屬離子輔因子——總計3個銅原子,1個鎂原子和1個鋅原子[39]。
這種酶承載了電子傳遞鏈的最終反應,在跨膜泵送質子時將電子轉移到氧上[40]。這一步,氧作為最終電子受體,也稱「末端電子受體」,被還原為水。直接泵送的質子和在氧的還原中消耗的基質中質子都能影響質子梯度。所催化的反應為細胞色素c的氧化及氧的還原:
替代的還原酶和氧化酶
[編輯]許多真核生物的電子傳遞鏈中的酶與上述研究較多的哺乳動物有所不同。例如,植物有替代的NADH氧化酶,可以不在粒線體基質而在細胞質中氧化NADH,並將這些電子傳遞到泛醌池[41]。這些酶不傳送子,可在不改變跨膜電化學梯度時還原泛醌[42]。
分岔電子傳遞鏈的另一個例子是「替代氧化酶」,存在於植物、一些真菌及原生生物中,一些動物也可能包含其中[43][44]。這種酶可直接將泛酚中的電子轉移到氧[45]。
通過這些替代NADH和泛醌氧化酶的電子傳遞途徑比完整路徑的ATP產量低。這些捷徑的優點尚不明確。然而,替代氧化酶產生於應激反應,如受寒、活性氧、病原體感染,及其他抑制完整電子傳遞鏈的因素[46][47]。因此,替代途徑可能可以通過減少氧化應激,從而提高生物對傷害的抵抗力[48]。
複合體組織
[編輯]最初的呼吸鏈複合體組織模型認為它們互相獨立,在粒線體膜中自由擴散[49]。然而,最近的資料表明,複合體可能形成稱為超複合體或「呼吸體」的高級結構[50]。在此模型中,各個複合體存在於有交互作用酶的有序集合中[51]。這些關聯可能使各種酶複合體之間建立了受質通道,增加電子轉移的速率和效率[52]。在哺乳動物的這些超複合體中,某些部件的數量可能較其它的要多,部分資料表明複合體I/II/III/IV和ATP合酶的比大致為1:1:3:7:4[53]。然而,對超複合體假說的爭論仍未完全結束,因為部分資料似乎無法支持此模型[17][54]。
原核生物的電子傳遞鏈
[編輯]雖然結構和功能與真核生物中的電子傳遞鏈相似,細菌和古菌的電子傳遞酶種類更為繁多。它們也擁有種類同樣繁多的化學物質作為受質[55]。與真核生物相同,原核生物的電子傳遞使用氧化受質所釋放的能量泵送離子跨膜,以產生電化學梯度。細菌中,對大腸桿菌的氧化磷酸化最為了解,而古菌中的這個過程目前仍知之甚少[56]。
真核生物和原核生物氧化磷酸化過程的主要區別是,細菌和古菌使用許多不同的物質以提供或接受電子。這使得原核生物可以在各種環境中生活[57]。例如在大腸桿菌中,許多還原劑和氧化劑對都可以驅動氧化磷酸化,如下所示。化學物質的中點電位可以衡量其被氧化或還原時所釋放的能量,其中還原劑具有負電位,氧化劑具有正電位。
| 呼吸酶 | 氧化還原對 | 中點電位(伏) |
|---|---|---|
| 甲酸去氫酶 | 碳酸氫鹽 / 甲酸鹽 | |
| 氫化酶 | 質子 / 氫 | |
| NADH去氫酶 | NAD+ / NADH | |
| 甘油-3-磷酸去氫酶 | DHAP / Gly-3-P | |
| 丙酮酸氧化酶 | 醋酸鹽 + 二氧化碳 / 丙酮酸鹽 | |
| 乳酸去氫酶 | 丙酮酸鹽 / 乳酸鹽 | |
| D-胺基酸去氫酶 | 2-含氧酸 + 氨 / D-胺基酸 | |
| 葡萄糖去氫酶 | 葡萄糖酸鹽 / 葡萄糖 | |
| 琥珀酸去氫酶 | 延胡索酸鹽 / 琥珀酸鹽 | |
| 泛酚氧化酶 | 氧 / 水 | |
| 硝酸鹽還原酶 | 硝酸鹽 / 亞硝酸鹽 | |
| 亞硝酸鹽還原酶 | 亞硝酸鹽 / 氨 | |
| 二甲基亞碸還原酶 | DMSO / DMS | |
| 氧化三甲胺還原酶 | TMAO / TMA | |
| 延胡索酸還原酶 | 延胡索酸鹽 / 琥珀酸鹽 |
如上所示,大腸桿菌可以在有還原劑如甲酸鹽、氫或乳酸作為電子供體,及硝酸鹽、DMSO、或氧氣作為受體的條件下生活[57]。氧化劑和還原劑之間的中點電位差異越大,它們發生反應時就能釋放更多的能量。這些化合物中,琥珀酸鹽/延胡索酸鹽對較特別,因為它的中點電位接近於零。琥珀酸鹽因此可以在有強氧化劑的條件下氧化成延胡索酸鹽,如氧,或者延胡索酸鹽在有強還原劑的條件下還原成琥珀酸鹽,如甲酸鹽。這些替代反應分別由琥珀酸去氫酶和延胡索酸還原酶催化[59]。
一些原核生物使用較小中間電位差的氧化還原對。例如,硝化細菌如硝化桿菌將亞硝酸鹽氧化為硝酸鹽,將電子傳給氧氣。反應中釋放的少量能量就足以泵送質子以產生ATP,但還不足以直接產生同化作用所需的NADH或NADPH[60]。通過使用亞硝酸鹽還原酶產生足夠的質子動力,讓電子傳遞鏈的一部分反向運行,使複合體I生成NADH,從而解決了此問題[61][62]。
原核生物可變換其產生的酶,控制電子供體和受體的使用,以適應環境條件的改變[63]。 因為不同的氧化酶和還原酶都使用相同的泛醌池,使其有一定的靈活性。由共同的泛醌中間體建立聯繫,酶的不同組合可以一起發揮作用[58]。呼吸鏈的模塊化設計使其擁有多套可交換的酶系統。
除了代謝的多樣性,原核生物也具有一系列的同工酶——可催化相同反應的不同酶。例如大腸桿菌有兩種不同類型的泛酚氧化酶,都用氧作為電子受體。在氧充足的條件下,細菌會使用氧親和性較低的氧化酶,其中每個電子可運送兩個質子。但如果氧含量下降,它就能換用每個電子只能運送一個質子,但對氧有高親合性的氧化酶[64]。
ATP合酶(複合體V)
[編輯]ATP合酶,又稱「複合體V」,是氧化磷酸化途徑中的終點酶。無論在原核生物還是真核生物中,這種酶的形式和作用方式都相同[65]。它用存儲在跨膜質子梯度的能量,驅動ADP和磷酸鹽(Pi)合成ATP。合成一個ATP分子所需的質子數估計為為3至4個[66][67],有人認為細胞可以調節這一比例以適應不同的條件[68]。
這個磷酸化反應是個可逆反應,可以通過改變質子動力變換反應方向。在不存在質子動力時,ATP合酶的反應將向左運行,水解ATP及將質子從基質中跨膜泵出。然而當存在高質子動力時,反應將被強制反向進行;反應從左向右進行,允許質子順濃度梯度流下,將ADP轉化為ATP[65]。事實上,與此密切相關的液胞型H +-ATP酶可通過泵送質子和水解ATP,從而酸化細胞區室[69]。
ATP合酶是一個巨大的蛋白質複合體,呈蘑菇狀。哺乳動物的酶複合體包含16個亞基,質量約為600千道爾頓[70]。嵌入在膜中的部分稱為FO,包含一個c亞基環和質子通道。柄和球形頭部稱為F1,是ATP的合成位點。F1端部的球形複合體包含兩種不同的六個蛋白(3個α亞基和3個β亞基),而「柄」包括一個蛋白:γ亞基,柄的前端延伸到球的α和β亞基[71]。α和β亞基都可與核苷酸結合,但只有β亞基可催化ATP的合成反應。與F1部分的一側接觸並延伸到膜背面的是一個長杆狀的亞基,將α和β亞基固定在酶的基底上。
當質子穿過ATP合酶基底的通道跨膜時,FO上的質子驅動馬達隨之旋轉[72]。轉動的原因可能是由於c亞基環上的胺基酸電離變化,引起靜電交互作用,從而推動c亞基環旋轉[73]。旋轉的環反過來驅動α和β亞基內的中心軸(γ亞基柄)旋轉。作為定子的α和β亞基被側臂固定以防止自身發生旋轉。在由α和β亞基組成的球內,γ亞基頂部的運動為β亞基的活性部位提供了能量,使其週期性地產生並釋放ATP[74]。

此ATP的合成反應稱為結合能力機制,其中涉及β亞基活性位點在三種狀態之間的循環[75]。在「空置」態,ADP和磷酸根進入活性位點(圖中棕色所示)。然後蛋白質包圍這些分子,並與之鬆散結合——是為「疏鬆結合」態(紅色所示)。隨後,酶再次改變形狀,迫使這些分子結合在一起,在隨後的「緊密結合」態(粉色所示)中,活性位點與新產生的極高親和力ATP分子結合。最後,活性位點循環回空置態,釋放出ATP並再次結合ADP和磷酸根,準備下一個循環。
在一些細菌和古菌中,ATP的合成是由鈉離子的跨細胞膜移動所驅動,而不是質子的移動[76][77]。古菌如甲烷球菌也包含A1Ao合酶,其包含額外的蛋白質,亞基順序與其他細菌和真核生物ATP合酶不相類似。也許在一些物種中,A1Ao的酶是一種特殊的鈉驅動ATP合酶[78],但可能在其他情況下並不總是正確的[77]。
活性氧物質
[編輯]氧分子是強氧化劑,因而是一種理想的末端電子受體。但氧的還原過程涉及有潛在危害的中間體[79]。雖然四個電子和四個質子的轉移而將氧還原為水的反應是無害的,一個或兩個電子的轉移會產生超氧或過氧陰離子,這是危險的反應。
這些活性氧和它們的反應產物,如羥基自由基,對細胞非常有害,因為它們能氧化蛋白質並導致DNA突變。細胞的損傷可能會誘發疾病,並可能是導致老化的原因之一[80][81]。
細胞色素c氧化酶複合體能高效地將氧還原為水,且只釋放極少量的部分還原中間體;然而,電子傳遞鏈卻會產生少量的超氧陰離子和過氧化物[82]。其中尤為重要的是輔酶Q在複合體III中的還原過程,因為作為一種中間體,高活性的泛半醌自由基會在Q循環中生成。這種不穩定的物質可能會導致電子的「洩漏」,從而直接將電子傳遞到氧,形成超氧化物[83]。由於在高膜電位時,這些質子泵複合物生成活性氧物質的速度最快,有人認為粒線體能調節自己的活動,使膜電位維持在一個狹窄的範圍內,以此平衡氧化劑和ATP的生成[84]。例如,氧化劑可以活化解偶聯蛋白,從而降低膜電位[85]。
為了清除活性氧,細胞有一套複雜的抗氧化劑體系,包括抗氧化維生素如維生素C和維生素E,抗氧化酶如超氧化物歧化酶、過氧化氫酶和過氧化物酶[79],它們能消除活性物質的毒性,減小對細胞的損傷。
抑制劑
[編輯]一些廣為人知的藥物和毒素可以抑制氧化磷酸化。儘管這些毒素的任何一個都只抑制電子傳遞鏈中的一個酶,抑制此過程中的任何步驟都將使其剩餘部分停止運轉。例如,如果寡黴素抑制了ATP合酶,質子就不能回流到粒線體內[86]。結果,質子泵就無法工作,因為梯度差太大以至於無法克服。然後,NADH不再被氧化,而因為NAD+濃度低於酶可用的濃度,三羧酸循環也隨之停止運轉。
| 化合物 | 作為 | 位置 | 效果 |
|---|---|---|---|
| 氰化物 一氧化碳 疊氮化物 硫化氫 |
毒素 | 複合體IV | 能比氧更強烈地結合細胞色素c氧化酶的鐵銅中心,阻止氧的還原,從而抑制電子傳遞鏈。[87] |
| 寡黴素 | 抗生素 | 複合體V | 阻止質子流過Fo亞基,從而抑制ATP合酶。[86] |
| CCCP 2,4-二硝基苯酚 |
毒素,減肥[e] | 內膜 | 攜帶質子跨膜的離子載體,從而破壞質子梯度。這種離子載體將質子泵與ATP合酶解耦,因為它能承載質子跨過粒線體內膜。[88] |
| 魚藤酮 | 農藥 | 複合體I | 阻斷與泛醌結合的位點,從而阻止電子從複合體I轉移到泛醌。[89] |
| 丙二酸鹽和草醯乙酸 | 毒素 | 複合體II | 琥珀酸去氫酶(複合體II)的競爭性抑制劑。[90] |
| 抗黴素A | 殺魚劑 | 複合體III | 結合到細胞色素C還原酶的Qi位點,從而抑制泛醌醇的氧化。 |
並非所有的氧化磷酸化抑制劑都是毒物。在棕色脂肪組織里,稱為解偶聯蛋白的調節性質子通道可將呼吸作用與ATP合成解耦[91]。它能使呼吸作用快速產生熱量,對冬眠動物和幼年哺乳動物來說,這對維持體溫尤為重要,雖然這些蛋白質可能在細胞的應激反應中有更一般化的功能[92]。
歷史
[編輯]對氧化磷酸化的研究起源於阿瑟·哈登1906年的報告,闡述了磷酸鹽在細胞發酵中的重要作用,但最初只知道糖磷酸鹽與此相關[93]。然而在20世紀40年代初,糖的氧化和ATP的生成之間的聯繫被赫爾曼·卡爾卡牢牢確立[94],同時在1941年,弗里茨·阿爾伯特·李普曼確認ATP在能量傳遞中起核心作用[95]。後來在1949年,莫里斯·弗里德金與阿爾伯特·倫寧格證明,輔酶NADH與代謝途徑如三羧酸循環及ATP的合成有關[96]。
又過了二十年,ATP的生成機制依然是個謎,同時科學家也在尋找那個難以捉摸的連接氧化與磷酸化反應的「高能中間體」[97]。這個難題在彼得·米切爾於1961年發表的化學滲透理論中得到了解決[98]。起初,這個看法極具爭議,但隨時間流逝,它逐漸為人們所接受,米切爾也於1978年獲頒諾貝爾化學獎[99][100]。隨後的研究集中於提純和描述所涉及的酶,其中戴維·格林和埃夫拉伊姆·萊克分別對電子傳遞鏈上複合體和ATP合酶的研究作出了重大貢獻[101]。解決ATP合酶機制的關鍵步驟由保羅·博耶1973年構想的「結合變構」機制所解釋,隨後是1982年他提出的旋轉催化的激進想法[75][102]。較近期的工作包括由約翰·沃克完成的氧化磷酸化酶的結構研究。沃克和博耶於1997年被授予諾貝爾化學獎[103]。
參見
[編輯]注釋
[編輯]參考文獻
[編輯]- ^ Mitchell P, Moyle J; Moyle. Chemiosmotic hypothesis of oxidative phosphorylation. Nature. 1967, 213 (5072): 137–9. Bibcode:1967Natur.213..137M. PMID 4291593. doi:10.1038/213137a0.
- ^ 2.0 2.1 Dimroth P, Kaim G, Matthey U. Crucial role of the membrane potential for ATP synthesis by F(1)F(o) ATP synthases. J. Exp. Biol. 1 January 2000, 203 (Pt 1): 51–9. PMID 10600673. (原始內容存檔於2007年9月30日).
- ^ 3.0 3.1 3.2 3.3 Schultz B, Chan S. Structures and proton-pumping strategies of mitochondrial respiratory enzymes. Annu Rev Biophys Biomol Struct. 2001, 30: 23–65. PMID 11340051. doi:10.1146/annurev.biophys.30.1.23.
- ^ Rich PR. The molecular machinery of Keilin's respiratory chain. Biochem. Soc. Trans. 2003, 31 (Pt 6): 1095–105 [2014-04-25]. PMID 14641005. doi:10.1042/BST0311095. (原始內容存檔於2019-09-12).
- ^ Porter RK, Brand MD. Mitochondrial proton conductance and H+/O ratio are independent of electron transport rate in isolated hepatocytes. Biochem. J. 1995, 310 ((Pt 2)): 379–82. PMC 1135905
 . PMID 7654171.
. PMID 7654171.
- ^ Mathews FS. The structure, function and evolution of cytochromes. Prog. Biophys. Mol. Biol. 1985, 45 (1): 1–56. PMID 3881803. doi:10.1016/0079-6107(85)90004-5.
- ^ Wood PM. Why do c-type cytochromes exist?. FEBS Lett. 1983, 164 (2): 223–6. PMID 6317447. doi:10.1016/0014-5793(83)80289-0.
- ^ Crane FL. Biochemical functions of coenzyme Q10. Journal of the American College of Nutrition. 1 December 2001, 20 (6): 591–8. PMID 11771674. doi:10.1080/07315724.2001.10719063. (原始內容存檔於2014年5月30日).
- ^ Mitchell P. Keilin's respiratory chain concept and its chemiosmotic consequences. Science. 1979, 206 (4423): 1148–59. Bibcode:1979Sci...206.1148M. PMID 388618. doi:10.1126/science.388618.
- ^ Søballe B, Poole RK. Microbial ubiquinones: multiple roles in respiration, gene regulation and oxidative stress management (PDF). Microbiology (Reading, Engl.). 1999, 145 (8): 1817–30. PMID 10463148. doi:10.1099/13500872-145-8-1817. (原始內容存檔 (PDF)於2008-05-29).
- ^ Johnson D, Dean D, Smith A, Johnson M. Structure, function, and formation of biological iron-sulfur clusters. Annu Rev Biochem. 2005, 74: 247–81. PMID 15952888. doi:10.1146/annurev.biochem.74.082803.133518.
- ^ Page CC, Moser CC, Chen X, Dutton PL; Moser; Chen; Dutton. Natural engineering principles of electron tunnelling in biological oxidation-reduction. Nature. 1999, 402 (6757): 47–52. Bibcode:1999Natur.402...47P. PMID 10573417. doi:10.1038/46972.
- ^ Leys D, Scrutton NS. Electrical circuitry in biology: emerging principles from protein structure. Current Opinion in Structural Biology. 2004, 14 (6): 642–7. PMID 15582386. doi:10.1016/j.sbi.2004.10.002.
- ^ Boxma B; de Graaf RM; van der Staay GW; Van Alen; Ricard; Gabaldón; Van Hoek; Moon-Van Der Staay; Koopman; Van Hellemond; Tielens; Friedrich; Veenhuis; Huynen; Hackstein; et al. An anaerobic mitochondrion that produces hydrogen. Nature. 2005, 434 (7029): 74–9. Bibcode:2005Natur.434...74B. PMID 15744302. doi:10.1038/nature03343.
- ^ 15.0 15.1 15.2 15.3 15.4 15.5 15.6 15.7 Medical CHEMISTRY Compendium. By Anders Overgaard Pedersen and Henning Nielsen. Aarhus University. 2008
- ^ 16.0 16.1 Hirst J. Energy transduction by respiratory complex I—an evaluation of current knowledge (PDF). Biochem. Soc. Trans. 2005, 33 (Pt 3): 525–9. PMID 15916556. doi:10.1042/BST0330525.
- ^ 17.0 17.1 Lenaz G, Fato R, Genova M, Bergamini C, Bianchi C, Biondi A. Mitochondrial Complex I: structural and functional aspects. Biochim Biophys Acta. 2006, 1757 (9–10): 1406–20. PMID 16828051. doi:10.1016/j.bbabio.2006.05.007.
- ^ 18.0 18.1 Sazanov, L.A.; Hinchliffe, P. Structure of the hydrophilic domain of respiratory complex I from Thermus thermophilus. Science. 2006, 311 (5766): 1430–1436. Bibcode:2006Sci...311.1430S. PMID 16469879. doi:10.1126/science.1123809.
- ^ Efremov R.G., Baradaran R., & Sazanov L.A., (2010) The arcdhitecture of respiratory complex I, Nature 465, 441-445
- ^ Baranova EA, Holt PJ, Sazanov LA. Projection structure of the membrane domain of Escherichia coli respiratory complex I at 8 A resolution. J. Mol. Biol. 2007, 366 (1): 140–54. PMID 17157874. doi:10.1016/j.jmb.2006.11.026.
- ^ Friedrich T, Böttcher B. The gross structure of the respiratory complex I: a Lego System. Biochim. Biophys. Acta. 2004, 1608 (1): 1–9. PMID 14741580. doi:10.1016/j.bbabio.2003.10.002.
- ^ Hirst J. Towards the molecular mechanism of respiratory complex I. Biochem. J. January 2010, 425 (2): 327–39. PMID 20025615. doi:10.1042/BJ20091382.
- ^ Cecchini G. Function and structure of complex II of the respiratory chain. Annu Rev Biochem. 2003, 72: 77–109. PMID 14527321. doi:10.1146/annurev.biochem.72.121801.161700.
- ^ Yankovskaya, V.; Horsefield, R.; Tornroth, S.; Luna-Chavez, C.; Miyoshi, H.; Leger, C.; Byrne, B.; Cecchini, G.; Iwata, S.; et al. Architecture of succinate dehydrogenase and reactive oxygen species generation. Science. 2003, 299 (5607): 700–704. Bibcode:2003Sci...299..700Y. PMID 12560550. doi:10.1126/science.1079605.
- ^ Horsefield R, Iwata S, Byrne B. Complex II from a structural perspective. Curr. Protein Pept. Sci. 2004, 5 (2): 107–18. PMID 15078221. doi:10.2174/1389203043486847.
- ^ Kita K, Hirawake H, Miyadera H, Amino H, Takeo S. Role of complex II in anaerobic respiration of the parasite mitochondria from Ascaris suum and Plasmodium falciparum. Biochim. Biophys. Acta. 2002, 1553 (1–2): 123–39. PMID 11803022. doi:10.1016/S0005-2728(01)00237-7.
- ^ Painter HJ, Morrisey JM, Mather MW, Vaidya AB; Morrisey; Mather; Vaidya. Specific role of mitochondrial electron transport in blood-stage Plasmodium falciparum. Nature. 2007, 446 (7131): 88–91. Bibcode:2007Natur.446...88P. PMID 17330044. doi:10.1038/nature05572.
- ^ Ramsay RR, Steenkamp DJ, Husain M. Reactions of electron-transfer flavoprotein and electron-transfer flavoprotein: ubiquinone oxidoreductase. Biochem. J. 1987, 241 (3): 883–92. PMC 1147643
 . PMID 3593226.
. PMID 3593226.
- ^ Zhang J, Frerman FE, Kim JJ; Frerman; Kim. Structure of electron transfer flavoprotein-ubiquinone oxidoreductase and electron transfer to the mitochondrial ubiquinone pool. Proc. Natl. Acad. Sci. U.S.A. 2006, 103 (44): 16212–7. Bibcode:2006PNAS..10316212Z. PMC 1637562
 . PMID 17050691. doi:10.1073/pnas.0604567103.
. PMID 17050691. doi:10.1073/pnas.0604567103.
- ^ Ikeda Y, Dabrowski C, Tanaka K. Separation and properties of five distinct acyl-CoA dehydrogenases from rat liver mitochondria. Identification of a new 2-methyl branched chain acyl-CoA dehydrogenase. J. Biol. Chem. 25 January 1983, 258 (2): 1066–76. PMID 6401712. (原始內容存檔於2007年9月29日).
- ^ Ruzicka FJ, Beinert H. A new iron-sulfur flavoprotein of the respiratory chain. A component of the fatty acid beta oxidation pathway (PDF). J. Biol. Chem. 1977, 252 (23): 8440–5. PMID 925004. (原始內容存檔 (PDF)於2007-09-27).
- ^ Ishizaki K, Larson TR, Schauer N, Fernie AR, Graham IA, Leaver CJ. The Critical Role of Arabidopsis Electron-Transfer Flavoprotein:Ubiquinone Oxidoreductase during Dark-Induced Starvation. Plant Cell. 2005, 17 (9): 2587–600. PMC 1197437
 . PMID 16055629. doi:10.1105/tpc.105.035162.
. PMID 16055629. doi:10.1105/tpc.105.035162.
- ^ Berry E, Guergova-Kuras M, Huang L, Crofts A. Structure and function of cytochrome bc complexes. Annu Rev Biochem. 2000, 69: 1005–75. PMID 10966481. doi:10.1146/annurev.biochem.69.1.1005.
- ^ Crofts AR. The cytochrome bc1 complex: function in the context of structure. Annu. Rev. Physiol. 2004, 66: 689–733. PMID 14977419. doi:10.1146/annurev.physiol.66.032102.150251.
- ^ Iwata S; Lee JW; Okada K; Kyongwon Lee; Iwata; Rasmussen; Link; Ramaswamy; Jap; et al. Complete structure of the 11-subunit bovine mitochondrial cytochrome bc1 complex. Science. 1998, 281 (5373): 64–71. Bibcode:1998Sci...281...64I. PMID 9651245. doi:10.1126/science.281.5373.64.
- ^ Trumpower BL. The protonmotive Q cycle. Energy transduction by coupling of proton translocation to electron transfer by the cytochrome bc1 complex (PDF). J. Biol. Chem. 1990, 265 (20): 11409–12. PMID 2164001. (原始內容存檔 (PDF)於2007-09-27).
- ^ Hunte C, Palsdottir H, Trumpower BL. Protonmotive pathways and mechanisms in the cytochrome bc1 complex. FEBS Lett. 2003, 545 (1): 39–46. PMID 12788490. doi:10.1016/S0014-5793(03)00391-0.
- ^ Calhoun M, Thomas J, Gennis R. The cytochrome oxidase superfamily of redox-driven proton pumps. Trends Biochem Sci. 1994, 19 (8): 325–30. PMID 7940677. doi:10.1016/0968-0004(94)90071-X.
- ^ Tsukihara T, Aoyama H, Yamashita E, Tomizaki T, Yamaguchi H, Shinzawa-Itoh K, Nakashima R, Yaono R, Yoshikawa S.; Aoyama; Yamashita; Tomizaki; Yamaguchi; Shinzawa-Itoh; Nakashima; Yaono; Yoshikawa. The whole structure of the 13-subunit oxidized cytochrome c oxidase at 2.8 A. Science. 1996, 272 (5265): 1136–44. Bibcode:1996Sci...272.1136T. PMID 8638158. doi:10.1126/science.272.5265.1136.
- ^ Yoshikawa S; Muramoto K; Shinzawa-Itoh K; et al. Proton pumping mechanism of bovine heart cytochrome c oxidase. Biochim. Biophys. Acta. 2006, 1757 (9–10): 1110–6. PMID 16904626. doi:10.1016/j.bbabio.2006.06.004.
- ^ Rasmusson AG, Soole KL, Elthon TE. Alternative NAD(P)H dehydrogenases of plant mitochondria. Annual review of plant biology. 2004, 55: 23–39. PMID 15725055. doi:10.1146/annurev.arplant.55.031903.141720.
- ^ Menz RI, Day DA. Purification and characterization of a 43-kDa rotenone-insensitive NADH dehydrogenase from plant mitochondria. J. Biol. Chem. 1996, 271 (38): 23117–20. PMID 8798503. doi:10.1074/jbc.271.38.23117. (原始內容存檔於2007-09-29).
- ^ McDonald A, Vanlerberghe G. Branched mitochondrial electron transport in the Animalia: presence of alternative oxidase in several animal phyla. IUBMB Life. 2004, 56 (6): 333–41. PMID 15370881. doi:10.1080/1521-6540400000876.
- ^ Sluse FE, Jarmuszkiewicz W. Alternative oxidase in the branched mitochondrial respiratory network: an overview on structure, function, regulation, and role. Braz. J. Med. Biol. Res. 1998, 31 (6): 733–47. PMID 9698817. doi:10.1590/S0100-879X1998000600003.
- ^ Moore AL, Siedow JN. The regulation and nature of the cyanide-resistant alternative oxidase of plant mitochondria. Biochim. Biophys. Acta. 1991, 1059 (2): 121–40. PMID 1883834. doi:10.1016/S0005-2728(05)80197-5.
- ^ Vanlerberghe GC, McIntosh L. Alternative oxidase: From Gene to Function. Annual Review of Plant Physiology and Plant Molecular Biology. 1997, 48: 703–34. PMID 15012279. doi:10.1146/annurev.arplant.48.1.703.
- ^ Ito Y, Saisho D, Nakazono M, Tsutsumi N, Hirai A. Transcript levels of tandem-arranged alternative oxidase genes in rice are increased by low temperature. Gene. 1997, 203 (2): 121–9. PMID 9426242. doi:10.1016/S0378-1119(97)00502-7.
- ^ Maxwell DP, Wang Y, McIntosh L; Wang; McIntosh. The alternative oxidase lowers mitochondrial reactive oxygen production in plant cells. Proc. Natl. Acad. Sci. U.S.A. 1999, 96 (14): 8271–6. Bibcode:1999PNAS...96.8271M. PMC 22224
 . PMID 10393984. doi:10.1073/pnas.96.14.8271. (原始內容存檔於2007-05-16).
. PMID 10393984. doi:10.1073/pnas.96.14.8271. (原始內容存檔於2007-05-16).
- ^ Lenaz G. A critical appraisal of the mitochondrial coenzyme Q pool. FEBS Lett. 2001, 509 (2): 151–5. PMID 11741580. doi:10.1016/S0014-5793(01)03172-6.
- ^ Heinemeyer J, Braun HP, Boekema EJ, Kouril R. A structural model of the cytochrome C reductase/oxidase supercomplex from yeast mitochondria. J. Biol. Chem. 2007, 282 (16): 12240–8. PMID 17322303. doi:10.1074/jbc.M610545200.
- ^ Schägger H, Pfeiffer K. Supercomplexes in the respiratory chains of yeast and mammalian mitochondria. EMBO J. 2000, 19 (8): 1777–83. PMC 302020
 . PMID 10775262. doi:10.1093/emboj/19.8.1777.
. PMID 10775262. doi:10.1093/emboj/19.8.1777.
- ^ Schägger H. Respiratory chain supercomplexes of mitochondria and bacteria. Biochim. Biophys. Acta. 2002, 1555 (1–3): 154–9. PMID 12206908. doi:10.1016/S0005-2728(02)00271-2.
- ^ Schägger H, Pfeiffer K. The ratio of oxidative phosphorylation complexes I-V in bovine heart mitochondria and the composition of respiratory chain supercomplexes. J. Biol. Chem. 2001, 276 (41): 37861–7. PMID 11483615. doi:10.1074/jbc.M106474200. (原始內容存檔於2007-09-29).
- ^ Gupte S; Wu ES; Hoechli L; Hoechli; Jacobson; Sowers; Hackenbrock; et al. Relationship between lateral diffusion, collision frequency, and electron transfer of mitochondrial inner membrane oxidation-reduction components. Proc. Natl. Acad. Sci. U.S.A. 1984, 81 (9): 2606–10. Bibcode:1984PNAS...81.2606G. PMC 345118
 . PMID 6326133. doi:10.1073/pnas.81.9.2606.
. PMID 6326133. doi:10.1073/pnas.81.9.2606.
- ^ Nealson KH. Post-Viking microbiology: new approaches, new data, new insights. Origins of life and evolution of the biosphere: the journal of the International Society for the Study of the Origin of Life. 1999, 29 (1): 73–93. PMID 11536899. doi:10.1023/A:1006515817767.
- ^ Schäfer G, Engelhard M, Müller V. Bioenergetics of the Archaea. Microbiol. Mol. Biol. Rev. 1999, 63 (3): 570–620. PMC 103747
 . PMID 10477309.
. PMID 10477309.
- ^ 57.0 57.1 Ingledew WJ, Poole RK. The respiratory chains of Escherichia coli. Microbiol. Rev. 1984, 48 (3): 222–71. PMC 373010
 . PMID 6387427.
. PMID 6387427.
- ^ 58.0 58.1 Unden G, Bongaerts J. Alternative respiratory pathways of Escherichia coli: energetics and transcriptional regulation in response to electron acceptors. Biochim. Biophys. Acta. 1997, 1320 (3): 217–34. PMID 9230919. doi:10.1016/S0005-2728(97)00034-0.
- ^ Cecchini G, Schröder I, Gunsalus RP, Maklashina E. Succinate dehydrogenase and fumarate reductase from Escherichia coli. Biochim. Biophys. Acta. 2002, 1553 (1–2): 140–57. PMID 11803023. doi:10.1016/S0005-2728(01)00238-9.
- ^ Freitag A, Bock E. Energy conservation in Nitrobacter. FEMS Microbiology Letters. 1990, 66 (1–3): 157–62. doi:10.1111/j.1574-6968.1990.tb03989.x.
- ^ Starkenburg SR; Chain PS; Sayavedra-Soto LA; et al. Genome Sequence of the Chemolithoautotrophic Nitrite-Oxidizing Bacterium Nitrobacter winogradskyi Nb-255. Appl. Environ. Microbiol. 2006, 72 (3): 2050–63. PMC 1393235
 . PMID 16517654. doi:10.1128/AEM.72.3.2050-2063.2006. (原始內容存檔於2007-10-13).
. PMID 16517654. doi:10.1128/AEM.72.3.2050-2063.2006. (原始內容存檔於2007-10-13).
- ^ Yamanaka T, Fukumori Y. The nitrite oxidizing system of Nitrobacter winogradskyi. FEMS Microbiol. Rev. 1988, 4 (4): 259–70. PMID 2856189.
- ^ Iuchi S, Lin EC. Adaptation of Escherichia coli to redox environments by gene expression. Mol. Microbiol. 1993, 9 (1): 9–15. PMID 8412675. doi:10.1111/j.1365-2958.1993.tb01664.x.
- ^ Calhoun MW, Oden KL, Gennis RB, de Mattos MJ, Neijssel OM. Energetic efficiency of Escherichia coli: effects of mutations in components of the aerobic respiratory chain (PDF). J. Bacteriol. 1993, 175 (10): 3020–5. PMC 204621
 . PMID 8491720. (原始內容存檔 (PDF)於2007-09-27).
. PMID 8491720. (原始內容存檔 (PDF)於2007-09-27).
- ^ 65.0 65.1 Boyer PD. The ATP synthase—a splendid molecular machine. Annu. Rev. Biochem. 1997, 66: 717–49. PMID 9242922. doi:10.1146/annurev.biochem.66.1.717.
- ^ Van Walraven HS, Strotmann H, Schwarz O, Rumberg B. The H+/ATP coupling ratio of the ATP synthase from thiol-modulated chloroplasts and two cyanobacterial strains is four. FEBS Lett. 1996, 379 (3): 309–13. PMID 8603713. doi:10.1016/0014-5793(95)01536-1.
- ^ Yoshida M, Muneyuki E, Hisabori T. ATP synthase—a marvellous rotary engine of the cell. Nature Reviews Molecular Cell Biology. 2001, 2 (9): 669–77. PMID 11533724. doi:10.1038/35089509.
- ^ Schemidt RA, Qu J, Williams JR, Brusilow WS. Effects of Carbon Source on Expression of Fo Genes and on the Stoichiometry of the c Subunit in the F1Fo ATPase of Escherichia coli. J. Bacteriol. 1998, 180 (12): 3205–8. PMC 107823
 . PMID 9620972.
. PMID 9620972.
- ^ Nelson N, Perzov N, Cohen A, Hagai K, Padler V, Nelson H. The cellular biology of proton-motive force generation by V-ATPases. J. Exp. Biol. 1 January 2000, 203 (Pt 1): 89–95. PMID 10600677. (原始內容存檔於2007年9月30日).
- ^ Rubinstein JL, Walker JE, Henderson R. Structure of the mitochondrial ATP synthase by electron cryomicroscopy. EMBO J. 2003, 22 (23): 6182–92. PMC 291849
 . PMID 14633978. doi:10.1093/emboj/cdg608.
. PMID 14633978. doi:10.1093/emboj/cdg608.
- ^ Leslie AG, Walker JE. Structural model of F1-ATPase and the implications for rotary catalysis. Philosophical Transactions of the Royal Society B. 2000, 355 (1396): 465–71. PMC 1692760
 . PMID 10836500. doi:10.1098/rstb.2000.0588.
. PMID 10836500. doi:10.1098/rstb.2000.0588.
- ^ Noji H, Yoshida M. The rotary machine in the cell, ATP synthase. J. Biol. Chem. 2001, 276 (3): 1665–8. PMID 11080505. doi:10.1074/jbc.R000021200. (原始內容存檔於2007-09-29).
- ^ Capaldi R, Aggeler R. Mechanism of the F(1)F(0)-type ATP synthase, a biological rotary motor. Trends Biochem Sci. 2002, 27 (3): 154–60. PMID 11893513. doi:10.1016/S0968-0004(01)02051-5.
- ^ Dimroth P, von Ballmoos C, Meier T. Catalytic and mechanical cycles in F-ATP synthases: Fourth in the Cycles Review Series. EMBO Reports. 2006, 7 (3): 276–82. PMC 1456893
 . PMID 16607397. doi:10.1038/sj.embor.7400646.
. PMID 16607397. doi:10.1038/sj.embor.7400646.
- ^ 75.0 75.1 Gresser MJ, Myers JA, Boyer PD. Catalytic site cooperativity of beef heart mitochondrial F1 adenosine triphosphatase. Correlations of initial velocity, bound intermediate, and oxygen exchange measurements with an alternating three-site model. J. Biol. Chem. 25 October 1982, 257 (20): 12030–8. PMID 6214554. (原始內容存檔於2007年9月29日).
- ^ Dimroth P. Bacterial sodium ion-coupled energetics. Antonie Van Leeuwenhoek. 1994, 65 (4): 381–95. PMID 7832594. doi:10.1007/BF00872221.
- ^ 77.0 77.1 Becher B, Müller V. Delta mu Na+ drives the synthesis of ATP via an delta mu Na(+)-translocating F1FO-ATP synthase in membrane vesicles of the archaeon Methanosarcina mazei Gö1. J. Bacteriol. 1994, 176 (9): 2543–50. PMC 205391
 . PMID 8169202.
. PMID 8169202.
- ^ Müller V. An exceptional variability in the motor of archaeal A1A0 ATPases: from multimeric to monomeric rotors comprising 6–13 ion binding sites. J. Bioenerg. Biomembr. 2004, 36 (1): 115–25. PMID 15168615. doi:10.1023/B:JOBB.0000019603.68282.04.
- ^ 79.0 79.1 Davies K. Oxidative stress: the paradox of aerobic life. Biochem Soc Symp. 1995, 61: 1–31. PMID 8660387.
- ^ Rattan SI. Theories of biological aging: genes, proteins, and free radicals. Free Radic. Res. 2006, 40 (12): 1230–8. PMID 17090411. doi:10.1080/10715760600911303.
- ^ Valko M, Leibfritz D, Moncol J, Cronin MT, Mazur M, Telser J. Free radicals and antioxidants in normal physiological functions and human disease. Int. J. Biochem. Cell Biol. 2007, 39 (1): 44–84. PMID 16978905. doi:10.1016/j.biocel.2006.07.001.
- ^ Raha S, Robinson B. Mitochondria, oxygen free radicals, disease and ageing. Trends Biochem Sci. 2000, 25 (10): 502–8. PMID 11050436. doi:10.1016/S0968-0004(00)01674-1.
- ^ Finkel T, Holbrook NJ. Oxidants, oxidative stress and the biology of ageing. Nature. 2000, 408 (6809): 239–47. PMID 11089981. doi:10.1038/35041687.
- ^ Kadenbach B, Ramzan R, Wen L, Vogt S. New extension of the Mitchell Theory for oxidative phosphorylation in mitochondria of living organisms. Biochim. Biophys. Acta. May 2009, 1800 (3): 205–212. PMID 19409964. doi:10.1016/j.bbagen.2009.04.019.
- ^ Echtay KS; Roussel D; St-Pierre J; Jekabsons; Cadenas; Stuart; Harper; Roebuck; Morrison; Pickering; Clapham; Brand; et al. Superoxide activates mitochondrial uncoupling proteins. Nature. January 2002, 415 (6867): 96–9. Bibcode:2002Natur.415...96E. PMID 11780125. doi:10.1038/415096a.
- ^ 86.0 86.1 Joshi S, Huang YG. ATP synthase complex from bovine heart mitochondria: the oligomycin sensitivity conferring protein is essential for dicyclohexyl carbodiimide-sensitive ATPase. Biochim. Biophys. Acta. 1991, 1067 (2): 255–8. PMID 1831660. doi:10.1016/0005-2736(91)90051-9.
- ^ Tsubaki M; Yoshikawa, Shinya. Fourier-transform infrared study of cyanide binding to the Fea3-CuB binuclear site of bovine heart cytochrome c oxidase: implication of the redox-linked conformational change at the binuclear site. Biochemistry. 1993, 32 (1): 164–73. PMID 8380331. doi:10.1021/bi00052a022.
- ^ Heytler PG. Uncouplers of oxidative phosphorylation. Meth. Enzymol. Methods in Enzymology. 1979, 55: 462–42. ISBN 978-0-12-181955-2. PMID 156853. doi:10.1016/0076-6879(79)55060-5.
- ^ Lambert AJ, Brand MD. Inhibitors of the quinone-binding site allow rapid superoxide production from mitochondrial NADH: ubiquinone oxidoreductase (complex I). J. Biol. Chem. 2004, 279 (38): 39414–20. PMID 15262965. doi:10.1074/jbc.M406576200. (原始內容存檔於2007-09-29).
- ^ Dervartanian DV, Veeger C. Studies on succinate dehydrogenase. I. Spectral properties of the purified enzyme and formation of enzyme-competitive inhibitor complexes. Biochim. Biophys. Acta. November 1964, 92: 233–47. PMID 14249115. doi:10.1016/0926-6569(64)90182-8.
- ^ Ricquier D, Bouillaud F. The uncoupling protein homologues: UCP1, UCP2, UCP3, StUCP and AtUCP. Biochem. J. 2000, 345 (2): 161–79. PMC 1220743
 . PMID 10620491. doi:10.1042/0264-6021:3450161.
. PMID 10620491. doi:10.1042/0264-6021:3450161.
- ^ Borecký J, Vercesi AE. Plant uncoupling mitochondrial protein and alternative oxidase: energy metabolism and stress. Biosci. Rep. 2005, 25 (3–4): 271–86. PMID 16283557. doi:10.1007/s10540-005-2889-2.
- ^ Harden A, Young WJ. The alcoholic ferment of yeast-juice. Proceedings of the Royal Society. 1906, B (77): 405–20. doi:10.1098/rspb.1906.0029.
- ^ Kalckar HM. Origins of the concept oxidative phosphorylation. Mol. Cell. Biochem. 1974, 5 (1–2): 55–63. PMID 4279328. doi:10.1007/BF01874172.
- ^ Lipmann F,. Metabolic generation and utilization of phosphate bond energy. Adv Enzymol. 1941, 1: 99–162.
- ^ Friedkin M, Lehninger AL. Esterification of inorganic phosphate coupled to electron transport between dihydrodiphosphopyridine nucleotide and oxygen. J. Biol. Chem. 1 April 1949, 178 (2): 611–23. PMID 18116985. (原始內容存檔於2008年12月16日).
- ^ Slater EC. Mechanism of Phosphorylation in the Respiratory Chain. Nature. 1953, 172 (4387): 975–8. Bibcode:1953Natur.172..975S. PMID 13111237. doi:10.1038/172975a0.
- ^ Mitchell P. Coupling of Phosphorylation to Electron and Hydrogen Transfer by a Chemi-Osmotic type of Mechanism. Nature. 1961, 191 (4784): 144–8. Bibcode:1961Natur.191..144M. PMID 13771349. doi:10.1038/191144a0.
- ^ Milton H. Saier Jr. Peter Mitchell and the Vital Force. [2014-05-25]. (原始內容存檔於2014-07-14).
- ^ Mitchell, Peter. David Keilin's Respiratory Chain Concept and Its Chemiosmotic Consequences (Pdf). Nobel lecture. Nobel Foundation. 1978 [2007-07-21]. (原始內容存檔 (PDF)於2007-09-27).
- ^ Pullman ME, Penefsky HS, Datta A, and Racker E. Partial Resolution of the Enzymes Catalyzing Oxidative Phosphorylation. I. Purification and Properties of Soluble, Dinitrophenol-stimulated Adenosine Triphosphatase. J. Biol. Chem. 1 November 1960, 235 (11): 3322–9. PMID 13738472. (原始內容存檔於2007年9月29日).
- ^ Boyer PD, Cross RL, Momsen W; Cross; Momsen. A New Concept for Energy Coupling in Oxidative Phosphorylation Based on a Molecular Explanation of the Oxygen Exchange Reactions. Proc. Natl. Acad. Sci. U.S.A. 1973, 70 (10): 2837–9. Bibcode:1973PNAS...70.2837B. PMC 427120
 . PMID 4517936. doi:10.1073/pnas.70.10.2837.
. PMID 4517936. doi:10.1073/pnas.70.10.2837.
- ^ The Nobel Prize in Chemistry 1997. Nobel Foundation. [2007-07-21]. (原始內容存檔於2017-03-24).
擴展閱讀
[編輯]概論
[編輯]- Nelson DL; Cox MM. Lehninger Principles of Biochemistry 4th. W. H. Freeman. 2004. ISBN 0-7167-4339-6.
- Schneider ED; Sagan D. Into the Cool: Energy Flow, Thermodynamics and Life 1st. University of Chicago Press. 2006. ISBN 0-226-73937-6.
- Lane N. Power, Sex, Suicide: Mitochondria and the Meaning of Life 1st. Oxford University Press, USA. 2006. ISBN 0-19-920564-7.
高級
[編輯]- Nicholls DG; Ferguson SJ. Bioenergetics 3 1st. Academic Press. 2002. ISBN 0-12-518121-3.
- Haynie D. Biological Thermodynamics 1st. Cambridge University Press. 2001. ISBN 0-521-79549-4.
- Rajan SS. Introduction to Bioenergetics 1st. Anmol. 2003. ISBN 81-261-1364-2.
- Wikstrom M (Ed). Biophysical and Structural Aspects of Bioenergetics 1st. Royal Society of Chemistry. 2005. ISBN 0-85404-346-2.
外部連結
[編輯]一般資料
[編輯]- 氧化磷酸化的動畫演示 (頁面存檔備份,存於網際網路檔案館),約翰威立《生物化學概念》
- 在線生物物理講座 (頁面存檔備份,存於網際網路檔案館),安東尼·克羅夫茨(Antony Crofts), 伊利諾伊大學厄巴納-香檳分校
- ATP合酶 (頁面存檔備份,存於網際網路檔案館),格雷厄姆·詹森(Graham Johnson)
- www.Atmungskette.de (精美顯淺易懂的呼吸鏈講解)
- Electron Transport Pathway--English (頁面存檔備份,存於網際網路檔案館)
分子結構資料
[編輯]- PDB的今月分子:
- 費爾南多·佩索阿大學的交互式分子模型:







